11.1. ВВЕДЕНИЕ
Во всем мире растет потребность в быстрых, точных и воспроизводимых методах идентификации микроорганизмов, ассоциируемых с мясом, мясом птицы и яйцепродуктами. Комитет экспертов по пищевой безопасности ФАО/ВОЗ констатировал еще в начале 1980-х гг., что пищевые отравления становятся самой распространенной проблемой здравоохранения и основной причиной снижения производительности труда. По разным оценкам в развитых странах ежегодно до 10% населения страдает от пищевых отравлений. В развивающихся странах ситуация намного хуже – там детская диарея зачастую обостряется и может привести к летальному исходу (см. http://z№W.cdcgov/mmm/previm/mrrm^ml/00018677.htm). Более того, перед органами здравоохранения в промышленно развитых странах в настоящее время остро стоит проблема новых или по-новому проявляющихся так называемых «эмерджентных» микрорганизмов пищевого происхождения. Некоторые из них способны вызывать серьезные заболевания, угрожающие жизни человека, особенно представителей групп повышенного риска, например пожилых людей и лиц с ослабленным иммунитетом, в том числе проходящим курс химиотерапии. В период 1988-1993 гг. число случаев заболевания сальмонеллезом в Австралии возросло вдвое, а в Японии – в 5 раз, так что основное внимание в настоящей главе мы уделим применяющимся в настоящее время методам ускоренной характеризации и идентификации микроорганизмов пищевого происхождения. Аналогичные подходы могут применяться и к микроорганизмам порчи пищевых продуктов, рассматривавшихся в главах 5 и 9, хотя в ходе стандартных исследований их редко оказывается возможным точно идентифицировать.
11.2. ВЫДЕЛЕНИЕ ИЗОЛЯТОВ В ЧИСТУЮ КУЛЬТУРУ
11.2.1. Состав ростовой среды
Для получения посевного материала для выполнения микробиологического исследования перед характеризацией и идентификацией какого-либо изолята его необходимо выделить в чистую культуру путем чашечного посева на неселективной агаровой среде, а затем вырастить в жидкой среде. Среда для выращивания микроорганизмов всегда должна содержать правильный баланс доступных им нутриентов. В зависимости от типа исследуемого микроорганизма в нее могут быть включены:
- соотвествующий источник азота (обычно используется пептон животного или растительного происхождения);
- дрожжевой, мясной или солодовый экстракт для обеспечения факторов роста, утраченных при изготовлении пептона, а также специфические аминокислоты;
- углеводы (обычно, глюкоза), обеспечивающие энергию;
- буфер, гарантирующий, что любые кислоты, продуцируемые в период роста, будут нейтрализованы и не воспрепятствуют дальнейшему росту микроорганизма.
11.2.2. Культивирование на агаровой среде
Для получения чистой культуры и проведения начальной характеризации неизвестного изолята обычно используют метод посева штрихом. Целью такого посева является распределение непрерывно уменьшающегося количества клеток по поверхности агара с последующим получением дискретных, хорошо отделенных друг от друга колоний. Данный метод позволяет увидеть характерные особенности колоний, что способствует процессу идентификации. В зависимости от состава культуральной среды можно наблюдать следующие характерные особенности одиночных колоний:
- форму и размер колоний;
- цвет (например, изменение значения рН);
- продуцирование пигментов;
- гемолиз, протеолиз, липолиз и т.д.
Один из наиболее популярных способов посева штрихом состоит в том, что берут петлю культуры и непрерывным волнистым движением распределяют ее на небольшом участке поверхности чашки по направлению от центра к краю. Затем другой стерильной петлей однократно проводят по площади, посеяной вначале, и извилистый штрих делают на соседнем небольшом участке чашки по направлению от центра к краю. Эти действия выполняют несколько раз до тех пор, пока не будет сделано пять отдельных штрихов так, чтобы гарантировалось отсутствие пересечения с любой зоной более обильного инокулята. Чашку инкубируют при температуре, соответствующей данному микроорганизму (зачастую при температуре, которая использовалась для исходного выделения). Такой способ должен дать очень хорошее разделение колоний. Чистая культура микроорганизма для последующей характеризации и идентификации может быть перенесена на агаровый косяк и подвергнута инкубации и хранению.
11.3. ХРАНЕНИЕ
11.3.1. Краткосрочное хранение
Большинство микроорганизмов, с которыми приходится иметь дело в пищевой промышленности, для определения микробного числа можно недолго поддерживать на косяках богатого питательными веществами агара двойной концентрации или косяках триптон-соевого агара (ТСА, TSA), которые хранят в охлажденном состоянии или при комнатной температуре. Вместе с тем, если культуры должны поддерживаться дольше чем несколько суток, их следует хранить иначе. Для таких энтеробактерий, как Salmonella, в США рекомендуется использовать основу кровяного агара, TSA или агар с сердечно-мозговой вытяжкой (см.: http://www.cdc.gov/ncidod/dbmd/ diseaseinfo/cholera/chlO.pdf). Такие углеводсодержащие среды, как агар Клиглера с цитратным железом или трехсахарный железосодержащий агар, использовать не рекомендуется, так как микроорганизмы будут превращать углеводы в кислоты, что впоследствии приведет к снижению жизнеспособности культуры.
Агаровые косяки приготавливают автоклавированием среды в пробирках и, пока она еще остается в жидком виде, наклоняют пробирки под углом 45° до затвердевания среды. Культуру берут с чашки, в которой она выделялась в чистую культуру, используя стерильную бактериальную петлю, и сначала погружают ее глубоко в пробирку с агаровым косяком (один или два раза), так что петля достигает торца пробирки, и затем делают штриховой посев на поверхности агара. Косяк инкубируют в течение ночи при соответствующей температуре. Пробирка должна быть плотно закрыта завинчивающейся крышкой или пробкой и обернута пленкой (парафильмом). Культуры следует хранить в темноте при комнатной температуре (22-25 °С). Для предотвращения высыхания культур можно использовать стерильное минеральное масло. Будучи приготовленной таким образом, культура остается жизнеспособной в течение многих месяцев и даже лет.
11.3.2. Долгосочное хранение
Долгое время для продолжительного хранения бактериальных культур использовалась лиофилизация (сублимационная сушка), однако большинству микробиологических лабораторий пищевых получить доступ к сублимационной сушке довольно трудно. С учетом этого был разработан современный и более простой метод, в котором используются пористые бусины. Для сохранения культуры этим способом хорошо изолированную колонию снимают стерильной петлей с чашки, где производилось выделение чистой культуры, и смешивают с бусинами в растворе для криогеного хранения. Затем бусины замораживают (желательно быстро), в морозильной установке с температурой –70 °С) и хранят в замороженном состоянии. В работе сообщается, что Clostridium difficile могут быть оживлены после хранения с использованием Protect Cryobeads™ (фирмы Hardy Diagnostics) в течение 1-2 лет, тогда как в традиционых условиях хранения большая часть изолятов не выживает.
11.4. ПРЕДВАРИТЕЛЬНОЕ ИССЛЕДОВАНИЕ
После того как на чашке с агаром выделен некий микроорганизм и получена его чистая культура, перед анализом необходимо провести ряд предварительных исследований (ориентировочное тестирование) в целях определения принадлежности данного микроорганизма к той или иной группе. Это имеет большое значение в связи с тем, что используемые в дальнейшем методы и процедуры для классификации и идентификации изолята существенно различаются в зависимости от реакции окрашивания по Граму, присутствия или отсутствия оксидазной и каталазной активности и способа диссимиляции глюкозы. Кроме того, следует учитывать и некоторые визуальные признаки, в зависимости от которых используют те или иные тесты. Примеры ориентироваочных тестов на основе анализа морфологии колоний или их реакций на чашках с агаром приведены ниже.
11.4.1. Характерные особенности колоний на чашке с агаром
Основные варианты морфологии колоний приведены на рис. 11.1. Кроме того, колонии микроорганизмов могут отличаться друг от друга:
- размерами (мм);
- цветом, например, быть белыми, желтыми, черными, палевыми, оранжевыми, оранжево-розоватыми;
- внешним видом поверхности: блестящей, матовой, иметь вид матового стекла;
- плотностью: быть непрозрачным, полупрозрачными или прозрачными;
- консистенцией, а именно маслянистыми, высоковязкими, пленчатыми, крошащимися, волосовидными.
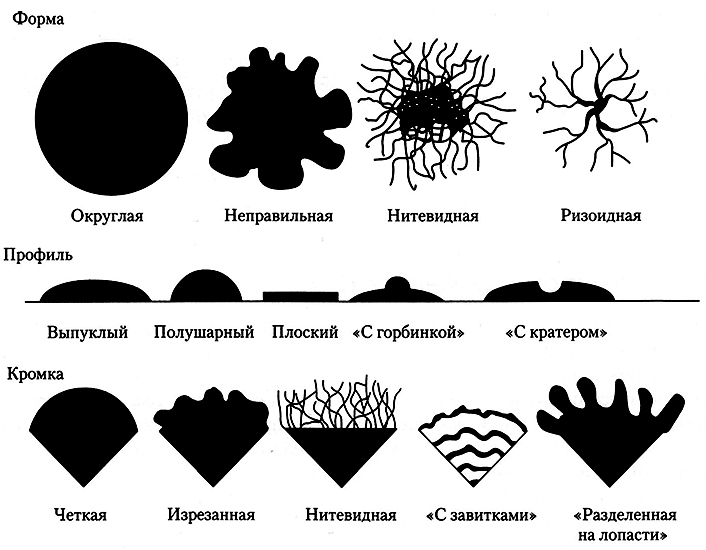
Рис. 11.1. Варианты морфологии колоний
11.4.2. Реакции, наблюдаемые на агаровой среде
Даже на неселективной агаровой среде типа питательного агара для определения микробного числа на чашках некоторые микроорганизмы продуцируют характерные пигменты. Примерами могут служить флуоресцентный пигмент, образуемый некоторыми псевдомондамами, сине-зеленый пигмент пиоцианин, образуемый Ps. aeroginosa и диффундирующий в агар, а также не способный к диффузии коричневый пигмент, продуцируемый некоторыми штаммами Bacillus subtilis и ограниченный пределами колонии. Иногда для обнаружения специфической ферментной реации можно использовать какой-либо субстрат. На желточном агаре, например, можно наблюдать несколько видов реакции – зону преципитации вокруг каждой колонии, образующуюся из-за действия лецитиназы, или радужный блеск на поверхности колоний («перламутровый слой») и узкую зону преципитации, образующиеся под действием липазной активности. Наоборот, зона просветления вокруг колонии свидетельствует о протеолизе.
Микроорганизм, растущий на специальной дифференцирующей среде, может характеризоваться определенными изменениями цвета под действием одного или нескольких побочных продуктов метаболизма, продуцируемых в процессе роста. Для проявления видимых эффектов, облегчающих идентификацию специфических микроорганизмов, к среде иногда добавляют красители, индикаторы рН и ферментативных реакций. О хромогенных средах см. также разделы 11.6.1 и 11.6.2.
11.4.3. Образование запаха
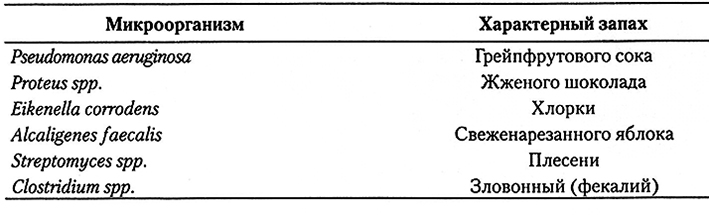
Многие штаммы микроорганизмов при росте в культуральной среде продуцируют характерные запахи, используемые при идентификации (см. табл. 11.1). Хотя только по запаху большинство микроорганизмов определить невозможно, разный запах, обусловленный продуцированием специфических метаболитов, может отражать различия между видами.
Таблица 11.1. Запахи, свойственные некоторым бактериям в культуре
11.4.4. Реакция окрашивания по Граму
Окрашивание по Грамму – это давно себя зарекомендовавший метод разделения бактерий на две большие группы по составу их клеточных стенок. В общих чертах он основан на способности некоторых микроорганизмов, в отличие от других, удерживать специфический краситель после процесса обесцвечивания. Бактерии, способные удерживать этот краситель, называют грамположительными, а остальные – грамотрицательными. Существует много разновидностей этого способа, но все они требуют много времени. Для ускорения анализа можно смешать колонию исследуемого микроорганизма с разбавленным раствором гидроокиси калия: образование гирлянды мылообразного материала является признаком грамотрицательного микроорганизма.
11.4.5. Проба на каталазу (каталазный тест)
Многие бактерии продуцируют фермент каталазу, что ведет к выделению кислорода из перекиси водорода. Для определения каталазной активности одну каплю 3%-ной перекиси водорода помещают непосредственно на колонию (или на мазок колонии на предметном стекле). Если микроорганизм каталазоположительный, то из-за образования пузырьков кислорода будет наблюдаться вспенивание.
11.4.6. Проба на цитохромоксидазу
Цитохромы являются ключевыми компонентами транспортных систем электронов. Для установления факта присутствия цитохромоксидазы культуру распределяют на бумажной полоске, пропитанной 1%-ным водным раствором тетраметил-n-фенилендиамина дигидрохлорида или оксалата. Быстрое появление синего окрашивания свидетельствует о положительном результате. Этот тест особенно полезен для различения грамотрицательных бактерий.
11.4.7. Способ утилизации глюкозы
Данный тест позволяет различать микроорагнизмы с двумя основными типами углеводного метаболизма. Производят посев микроорганизма уколом в две пробирки с агаровой средой, содержащей глюкозу и индикатор рН. Одну пробирку инкубируют на воздухе, другую – анаэробно или герметизируют агаром. Микроорганизмы с окислительным типом метаболизма будут утилизировать глюкозу только в присутствии воздуха, а ферментирующие микроорганизмы дадут положительный результат в обеих пробирках. Некоторые микроорганизмы не утилизируют углеводы в качестве источников энергии, и в этом случае обе пробирки дадут отрицательный результат.
11.5. СИСТЕМЫ ИДЕНТИФИКАЦИИ МИКРООРГАНИЗМОВ
11.5.1. Миниатюризированные тест-наборы
Традиционные методы идентификации патогенных бактерий, а также тех бактерий, дрожжей и плесеней, что вызывают порчу пищевых продуктов, очень трудоемки и требуют много времени. Так, для обнаружения и идентификации патогенных бактерий рода Salmonella с использованием традиционных методов может потребоваться до 7 сут, а определение численности психротрофных микроорганизмов посевом на чашках Петри (даже без попыток идентифицировать какой-либо из них) требует инкубационного периода в 10 сут при температуре 7 °С. Поскольку такие скоропортящиеся продукты, как мясо птицы, должны поступать на реализацию не позднее, чем через 24 ч после переработки, то они, по всей вероятности, будут употреблены в пищу задолго до получения результатов анализов. Все это привело к появлению многочисленных миниатюризированных тест-наборов и тест-систем для экспресс-диагностики, некоторые из которых мы рассмотрим ниже.
Тест-наборы API®
Среди различных биохимических наборов фирмы bioMerieux тест-наборы API® специально предназначены для идентификации следующих микроорганизмов: API20А – для анаэробных, API CAMPY– для Campylobacterspp., Rapid CORYNE – для corynebacteria, API 20E – для представителей семейства Enterobacteriaceae, API NFT – для грамотрицательных бактерий, не относящихся к семейству Enterobacteriaceae, API 50СН – Lactobacillus spp., API LISTERIA – для Listeria spp., API Staph-IDENT – для Staphylococcus и Micrococcus spp., Rapid STREP – для Streptococcus spp. и API 20C – для дрожжей. Для использования любого из этих тест-наборов готовят суспензию исследуемого микроорганизма в солевом растворе и наносят эту суспензию на стрип, содержащий различные биохимические субстраты для ассимиляционных тестов. Затем стрип инкубируют при заданной температуре заданное время. После инкубации результаты тестов регистрируют либо вручную, либо данные со стрипа считаываются автоматически. Иногда для проявления результата требуется дополнительное внесение специальных реактивов. Для получения результата в цифровом виде (то есть в виде кода культуры микроорганизма), используется вся последовательность реакций на стрипе. Идентификация осуществляется путем сопоставления полученного кода с базой данных.
В работе сообщается, что при сравнении с традиционными методами система API 20Е правильно идентифицирует соответствующие микроорганизмы до уровня рода и вида в 90,2% случаев. Утверждается, что преимущества этого тест-набора, в отличие от других, состоят в том, что тесты легко засевать и учитывать, что доступна обширная база данных и что результаты получаются относительно быстро. К ее недостаткам можно отнести то, что тест-стрипы трудно устанавливать в термостате и манипулировать с ними, а результаты не всегда легко интерпретировать.
Тест-система Micro-ID®
Тест-система Micro-ID" (фирмы Remel Inc.) предназначена для экспресс-идентификации бактерий семейства Enterobacteriaceae. Каждый тест-стрип содержит 20 дисков из фильтровальной бумаги, пропитанной реактивами. Посевной материал исследуемой бактерии распределяется по рядам лунок, в каждой из которых находится один диск. Реактивы, присутствующие на диске, включают субстрат, который реагирует с бактериальным ферментом, и такую систему обнаружения продукта(ов) метаболизма, которая позволяет легко распознавать изменение цвета за 4 ч инкубации. Для получения цифрового кода и последующего сопоставления его с базой данных для идентификации микроорганизма используется последовательность реакций.
Отмечается, что при сравнении с традиционными методами система Micro-ID® успешно идентифицирует микроорганизмы до уровня рода и вида в 93,5% случаев. Эта система по сравнению с другими легче для засева, требует более короткого времени инкубации, меньше времени для добавления реагентов и относительно проста для учета результатов. Недостатком можно считать то, что результаты не всегда легко интерпретировать.
Тест-системы IDS RapID™
Для идентификации микроорганизмов с помошью систем RapID™ (фирмы Remel Inc.) колонию каждого исследуемого микроорганизма смешивают с посевной средой и получают суспензию необходимой плотности. Эту суспензию затем используют для посева на тест-стрип с 10-18 субстратами (в зависимости от типа идентифицируемых бактерий или дрожжей). При добавлении посевного материала дегидратированные реактивы, находящиеся в лунках, быстро регидратируются. При утилизации некоторых из субстратов тестовым микроорганизмом происходит изменение цвета. Получающаяся в результате последовательность (профиль) положительных и отрицательных реакций сравнивается с последовательностью из базы данных. Коммерчески доступны стрипы для идентификации следующих неферментирующих представителей семейства Enterobacteriaceae: Haemophilus spp., Leuconostoc spp., Listeria spp., Neisseria spp., Pediococcus spp. и Streptococcus spp., а такде для анаэробных бактерий, бактерий мочевых путей и дрожжей.
Эффективность системы RapID ANA 11 для идентификации анаэробных микроорганизмов исследовалась в работе. Точность идентификации штаммов до уровня рода составила для грамотрицательных палочек 96%, для Clostridium spp. – 94%, для неспорообразующих грамположительных палочек – 83%, а для кокков – 97%. До уровня вида правильно были идентифицированы соответственно 86,76,81 и 97% микроорганизмов.
Система Biolog
В системе идентификации микроорганизмов Biolog (фирмы Biolog) используются 96-луночные микротитрационные платы, в которые предварительно загружены реагенты для метаболических тестов. Каждая лунка в ряду содержит разный источник углерода и индикаторную систему определения редокс-потенциала. Сухие химикаты регидратируются при внесении бактериальной клеточной суспензии определенной мутности. Во время инкубации бактерия утилизирует источники углевода в некоторых из лунок. Если утилизируется специфический компонент, то происходит окислительно-восстановительная реакция и окраска среды меняется в течение 4-24 ч от бесцветной до пурпурной. Изменение окраски может определяться визуально или с помощью считывающего устройства для микроплат, соединенного с компьютером (ридера). После считывания информации программное обеспечение MicroLogrM преобразует полученную информацию в цифровой код, который сопоставляется с базой данных. Эта система может использоваться для идентификации многих видов грамположительных и грамотрицательных бактерий. Кроме того, разработаны микроплаты для идентификации Escherichia и Salmonella spp.
В фирме Biolog разработаны также некоторые 96-луночные платы для микротитрования МТ Micro Plates™, содержащие ту же питательную основу и цветообразующие реагенты, что и раньше, но без источников углерода. Эти микроплаты могут быть приспособлены для специальных целей путем добавления различных источников углерода, что позволяет расширить потенциальные метаболические возможности. Оценка способности системы Biolog дифференцировать различные бактериальные изоляты дала следующие результаты: из 39 протестированных в течение 4-24 ч штаммов из American Type Culture Collection 98% были идентифицированы правильно до уровня рода и 76% – до уровня вида. В целом эту систему легко использовать, она относительно недорога и может идентифицировать много различных штаммов, однако для получения непротиворечивых результатов важно, чтобы микротитрационная плата была правильно засеяна, а это требует наличия практического навыка. Чтобы избежать переноса питательного материала тампоном для взятия колонии, лаборант должен быть очень внимательным, так как этот материал может попасть в реакционные лунки, поддержать рост тестового микроорганизма и вызвать ложноположительное изменение цвета, вследствие чего микроорганизм может быть идентифицирован неправильно или вообще не идентифицирован.
Тест-система BBL™ Crystal™
Эта система, предлагаемая фирмой Becton-Dickinson Microbiology Systems, представляет собой относительно новую систему идентификации бактерий. Существуют два тест-набора на основе этой системы: Rapid Stool/Enteric ID Kit (RS/E Kit) и Enteric/Nonfermenter ID Kit (E/NF Kit). В них использованы модифицированные традиционные и хромогенные субстраты (всего 30) в платах нового типа. Каждый тест-набор состоит из пластикового основания с реакционными лунками, на которое после посева прикрепляют крышку с дегидратированными субстратами, помещенными на концы пластиковых зубцов. После инкубации в течение 3 ч (для RS/E Kit) и 18-20 ч (для E/NF Kit) результаты интерпретируют визуально и регистрируют вручную. Затем их преобразуют в 10-значный цифровой код, который сравнивают с базой данных и тем самым идентифицируют микроорганизм.
По сравнению с системами API® и VITEK® (см. ниже) система Crystal™ характеризуется хорошей эффективностью. Сообщается, что с её помощью из 512 грамотрицательных палочек (381 – представители семейства Enterobacteriaceae и 131 – «неэнтеробактерии») 95,5% были идентифицированы точно до уровня рода и вида. Фактически 93,9% штаммов были идентифицированы в течение 24 ч без какой-либо необходимости дополнительного тестирования. Ошибки идентификации при анализе с помощью этой системы были редки и характеризовались случайным распределением.
Тест-наборы RS/E и E/NF оцеивали также и по их способности идентифицировать грамотрицательные микроорганизмы. Так, из проанализированных соответственно 203 и 266 штаммов было правильно идентифицировано 91 и 93% бактерий семейства Enterobacteriaceae. Эти системы безопасны и просты в использовании.
При сравнении этих миниатюризованных тест-наборов для идентификации микроорганизмов с традиционными методами анализа оказалось, что они относительно точны, требуют меньше места для инкубации, не требуют времени и труда для приготовления сред, а результаты получаются намного быстрее. Кроме того, консистенция материалов, используемых в этих тест-наборах, очень удобна, причем практически исключены ошибки, связанные с неправильным приготовлением или стерилизацией традиционных сред. Тем не менее при использовании таких тест-наборов существует опасность ошибок при посеве, так как необходимо использовать определенную концентрацию клеток. Следует отметить, что культуры, которые не эмульгируются в солевом растворе, идентифицировать с помощью данных систем трудно, а в некоторых случаях и невозможно.
11.6. НАИБОЛЕЕ РАСПРОСТРАНЕННЫЕ
СЕЛЕКТИВНЫЕ И ДИФФЕРЕНЦИРУЮЩИЕ СРЕДЫ
В данном разделе мы рассмотрим среды, позволяющие предположительно идентифицировать выделенные микроорганизмы и почти не требующие применения подтверждающих анализов.
11.6.1. Способы обнаружения
микроорганизмов-индикаторов фекального загрязнения
Petrifilm™
Метод, используемый для выявления бактерий на поверхностях оборудования и т.д., включает использование пленок сухой питательной среды, поставляемых компанией ЗА/. Базовая система состоит из двух прилегающих друг к другу пленок – одна с питательными веществами, принятыми в стандартных методах (SM), а другая – с растворимым в холодной воде гелеобразующим веществом. Нижняя пленка покрыта 5М-питательными компонентами, а верхняя пленка – гелеобразующим агентом плюс трифенилтетразолиум хлоридом, благодаря чему подсчет колоний облегчается окрашиванием колоний в красный цвет.
Для использования Petrifilm™ для определения численности микроорганизмов путем прямого контакта 1 мл стерильной 0,1%-ной пептонной воды помещают на бумагу, покрывающую пленку, и дают возможность пленке со средой контактировать с жидкостью в течение 30 мин. Затем пленку удаляют, и гидратированную среду используют для взятия пробы путем прижимания ее к исследуемой поверхности. Среду после этого инкубируют, и число колоний выражают в виде количества КОЕ на 10 см2. Популяции колиформ и Е. coli можно оценить соответственно с помощью систем Petrifilm Coliform Count Plates и E. coli Count Plates. По сравнению с традиционными методами определения численности микроорганизмов посевом на чашки, этот способ относительно недорог, требует меньше трудозатрат, а результаты могут быть получены вместо 48 ч в течение суток. К его недостаткам можно отнести то, что пептонную воду необходимо приготавливать отдельно, среда на пленке имеет тенденцию размазываться, а другими методами результаты можно получить намного быстрее.
Хромогенные среды
В последнее время возросла популярность хромогеных сред как средств различения разных микроорганизмов, позволяющих предположительно идентифицировать некоторые группы микроорганизмов, ассоциируемых с пищевыми продуктами. Дифференциация в данном случае основывается на изменении цвета среды, при реакции продуктов микробного метаболизма с хромогенными веществами среды. Примерами могут служить среды CPS ID2 (фирма bioMerieux) и CHROMagar™ (СМО, фирма Dr A Rambach – BBL™ CHROMagar™ Orientation). Эти среды представляют собой твердые субстраты в готовой для использования форме, которые служат для одноэтапного выделения и идентификации Е. coli и Enterococcus spp. В них содержатся два различных хромогенных субстрата: CPS ID2 позволяет определять p-глюкуронидазу и индол, продуцируемые Е. coli (синий цвет), триптофандезаминазу, продуцируемую Proteus (коричневый цвет), и β-глюкозидазу, продуцируемую энтерококками. На среде СМО можно определять ферменты, ассоциируемые с метаболизмом лактозы, в результате чего получается окрашивание от розового до красного. Колонии, вырастающие на этих средах, бывают окрашены в розовый, красный, голубой, сине-зеленый или пурпурный цвета (если они не продуцируют соответствующий фермент, то их природный цвет не меняется). Эти две среды изначально предназначались для использования в качестве неселективных сред для выделения, количественного определения и дифференцирования патогенных бактерий мочевыводящих путей, однако в настоящее время они применяются для обнаружения Е. coli и Enterococcus spp. в пробах пищевых продуктов и не требуют подтверждающих анализов.
Большинство исследований с использованием этих сред было выполнено на образцах мочи. В работе, например, на средах CPS ID2 и СМО было выделено 266 штаммов Е. coli из образцов, тогда как на кровяном агаре было выделено только 260 штаммов, а на агаре МакКонки – 248 штаммов. Один штамм (0,4%) не дал ожидаемого цвета на среде СМО, а 23 штамма (8,7%) не образовали его и на средеCPS ID2. Кроме того, были выделены 266 штаммов энтерококков, которые на обеих средах давали мелкие сине-зеленые колонии. И в том и в другом случае можно было идентифицировать группы бактерий Klebsiella-Enterobacter-Serratia (KES) и Proteus-Morganella-Providencia. Таким образом, 58 из 64 KES-штшоъ на среде CPS ID2 дали ожидаемый цвет, а на среде СМО – 63 из 66 штаммов. Это позволяет прийти к выводу, что одним из преимуществ этих сред является легкость распознавания конкретных микроорганизмов в смешанных популяциях бактерий.
В работе методом многоточечной инокуляции была проведена оценка среды СМО относительно возможностей дифференциации и предположительной идентификации грамположительных палочек и Enterococcus spp. Протестированные штаммы включали 1404 штамма грамотрицательных палочек и 74 штамма энтерококков. Было установлено, что розово-красные колонии продуцируют 99,3% штаммов Е. coli. Вместе с тем оказалось, что четыре штамма Е. coli характеризуются отрицательной реакцией на о-нитрофенил- β-D-галактопиранозид, в связи с чем их цвет не менялся. Штаммы Proteus mirabilis и P. vulgaris на среде СМО легко различимы. Штаммы P. mirabilis (n = 184) продуцируют прозрачные колонии с диффундирующим коричневым пигментом по периферии, а штаммы P. vulgaris дают голубовато-зеленую окраску с легким коричневым фоном. Все 26 штаммов Aeromonas hydrophila после инкубации при температуре 35-37 °С образовывали колонии (от прозрачных до розовых). С другой стороны, при комнатной температуре цвет колоний изменялся на голубой уже через 2-3 ч. Колонии Serratia marcescens (n - 29) имели цвет морской волны, который при комнатной температуре становился темнее. Все 74 штамма энтерококков давали синий цвет, но не отличались от Klebsiella, Enterobacter и Citrobacter spp., хотя эти микроорганизмы можно было легко отличить от других Enterobacteriaceae. В результате был сделан вывод, что среда СМО позволяет легко различать некоторые микроорганизмы в смешанной микробиоте.
При изучении образцов мочи пробы параллельно сеяли на чашки с агаром, содержащим овечью кровь, на чашки с агаром МакКонки и СМО, после чего сравнивали результаты после инкубации. В большинстве случаев между указанными тремя средами наблюдалось полное соответствие. То же самое имело место при анализе 400 культур одиночного патогена и девяти смешанных культур на численность выделенных микроорганизмов и их идентичность. Использование среды СМО сократило продолжительность инокуляции более чем на 50%, а анализа – более чем на 20%.
11.6.2. Хромогенные среды
для обнаружения патогенных микроорганизмов
Для некоторых основных пищевых патогенов разработаны хромогенные среды, облегчившие выделение и дифференцирование этих микрорганизмов. Некоторые из них аттестованы Международной ассоциацией химиков-аналитиков (АОАСГ) для микробиологического исследования определенных пищевых продуктов при использовании в качестве неотъемлемой части официального метода анализа. Примером тому является среда CHROMagar™ О157 (СМА О157), дифференцирующая Е. coli О157 от других штаммов Е. coli с помощью высоко специфичного хромогенного субстрата для β-глюкуронидазы. На этой среде штаммы серотипа О157 дают розовато-лиловые окрашенные колонии, а другие грамотрицательные бактерии подавляются или дают бесцветные, голубые, зеленые и сине-зеленые колонии. Эффективность этой среды исследовалась в работе, и было показано, что с ее помощью можно сократить время определения положительных образцов, а также затраты труда и материалов, требуемых для подтверждения идентичности целевого микроорганизма.
При первичном выделении трудно различить Listeria monocytogenes от других Listeria spp., но это можно сделать с использованием агара RAPID'L mono™ (фирмы Biorad), так как L. monocytogenes продуцируют фермент фосфатидилинозитолфосфолипазу С (PIPLC), и, кроме того, этот микроорганизм не может метаболизировать присутствующую в среде ксилозу. Единственными представителями Listeria spp. с PIPLC-активностью являются L. monocytogenes и L. ivanovii. Включение ксилозы в среду позволяет идентифицировать их друг от друга. L. ivanovii, утилизирующие ксилозу, образуют колонии с отчетливыми желтыми гало, a L. monocytogenes – голубые колонии без таких гало. Остальные непатогенные листерии дают белые колонии. Применение агара RAPID'L mono™ позволяет проводить предположительную идентификацию L. monocytogenes в течение 24 ч, однако степень обнаружения других листерий довольно низка, так как в течение 7-суточного инкубационного периода не все штаммы оказались способны расти или проявлять P/PZC-активность на этой среде.
Еще одной хромогенной средой для обнаружения листерий является CHROMagar™ Listeria (CMAL), на которой целевые микроорганизмы при засеве из первичного обогатительного бульона дают бирюзовые колонии за 24 ч при температуре 36 ± 1 °С. Это оказывается возможным благодаря хромогенному субстрату на основе 5-бром-4-хлор-3-индоксил- β-D-глюкопиранозида, позволяющего обнаруживать β-D-глюкозидазную активность, наблюдающуюся у всех видов листерий. Для распознавания патогенных L. monocytogenes и L. ivanovii в среде используется расщепление L-фосфатидилинозитола с помощью PIPLC и образование вокруг каждой колонии зоны белой преципитации. Поставщик (фирма BBL) утверждает, что среда CMAL характеризуется чувствительностью 99-100% и 100%-ной специфичностью.
Штаммы Staphylococcus aureus можно выделять с помощью новой системы микробиологического анализа Petrifilm™ Staph Express Count Plate (фирмы 3M). Эта тест-система содержит водорастворимый гелеобразующий агент и хромогенную модифицированную среду Бэйд-Паркера, являющуюся для данного целевого микроорганизма селективной и дифференцирующей. Предполагаемые колонии имеют красно-фиолетовый цвет, а поскольку Staph, aureus может расщеплять ДНК, которая вместе с соответствующим красителем включена в состав среды, то колонии этого микроорганизма окружены розовыми зонами. Когда мясо птицы механической обвалки тестировали с помощью системы Petrifilm™ и традиционной среды Бэйд-Паркера, результаты оказались сопоставимы, как и в случае других пищевых продуктов животного происхождения.
Среда BBL™ CHROMagar™ Staph, aureus (CSA) представляет собой пример еще одной хромогенной среды, на которой Staph, aureus образует колонии розоватолилового цвета, однако для их подтверждения необходимо провести тест на коагулазу. В случае клинических образцов суммарная чувствительность составила 99,5%, а специфичность – 98%.
Что касается сальмонелл, то зачастую бывает трудно отличить предполагаемые колонии от колоний конкурирующих микроорганизмов. Тем не менее на среде BBL™ CHROMagar™ Salmonella розово-фиолетовые колонии легко заметны среди других бактерий, включая колиформы, имеющие сине-зеленые, либо бесцветные колонии. По сравнению с традиционными средами для выделения время, необходимое для подтверждения предполагаемых колоний, сократилось до 24 ч.
Предлагаются также хромогенные среды для выявления дрожжей и мицелиальных грибов, а также для дифференциации Candida spp. Например, в состав среды BBL™ CHROMagar™ Candida включен хромогенный субстрат, позволяющий С. albicans, С. tropicalis и С. krusei образовывать колонии разного цвета – соответственно, от светло-зеленого до зеленого, от темно-синего до металлического синего и от светло-розового до розовато-лилового. Эти колонии, кроме того, имеют плоскую форму с беловатой кромкой. Колонии остальных дрожжей по сравнению с ними будут выглядеть или светлее, или темнее.
11.7. АВТОМАТИЗИРОВАННАЯ
ИДЕНТИФИКАЦИЯ МИКРООРГАНИЗМОВ
Уже довольно давно известны и применяются автоматизированные аналитические системы идентификации микроорганизмов на основе данных по утилизации субстрата, анализа клеточных жирных кислот (табл. 11.2), а также метаболических продуктов микроорганизмов на основе газовой хроматографии.
Таблица 11.2.
Автоматические системы для идентификации микроорганизмов

11.7.1. Системы, основанные на анализе
утилизации субстрата VITEK9
Автоматизированная микроробиологическая система VITEK AutoMicrobic System (фирм AMS, bioMerieux) является одним из наиболее универсальных способов ускоренной идентификации микроорганизмов. Для идентификации изолята колонию микроорганизмов смешивают в пробирке с 0,45%-ным солевым раствором, получая суспензию определенной мутности в зависимости от типа подлежащего идентификации микроорганизма. Эта суспензия используется затем для засева прозрачной карты. Один конец маленькой пластиковой трубочки вставляют в тест-карту, а другой конец помещают в находящуюся в пробирке суспензию. Карту и пробирку с суспензией помещают в штатив устройства для заполнения карт. Штатив затем переносят в устройство для заполнения, в котором для вытягивания суспензии из пробирки в каждую из 30 тест-ячеек карты со специфическими компонентами используется вакуум. Карта герметично укупоривается, вставляется в держатель и помещается в автоматизированный анализатор.
По мере утилизации исследуемым микроорганизмом определенных субстратов из ячеек карты оптическая плотность каждой реакционной смеси меняется. Измерение оптической плотности выполняется 1 раз в час, причем в целях идентификации микроорганизмов получаемые сведения сравниваются со стандартной базой данных. Обычно изоляты удается идентифицировать в течение 4-18 ч, причем одновременно можно анализировать до 120 проб.
Предлагается 8 различных тест-карт в зависимости от идентифицируемых микроорганизмов: для грамотрицательных (GNI), грамположительных (GPI), неферментирующих (NFC), дрожжей ( YBC), бацилл (ВАС), анаэробов (ANI), для количественного определения бионагрузки (Bioburden Enumeration, BIO) и аналитическая карта (Assay Card, ASC). BIO-карту используют для определения численности микробной популяции в жидких образцах, а ASC-карту – для измерения концентрации или эффективности антибиотиков, витаминов, биоцидов или консервантов путем мониторинга роста микроорганизмов в присутствии соответствующих веществ.
Эффективность автоматического анализатора (AMS) при идентификации хранившихся культур и свежевыделенных штаммов Enterobacteriaceae из говяжьего фарша, различных продуктов из куриного мяса и кормов для птицы изучалась в работе. Оказалось, что данная тест-система правильно идентифицировала до уровня вида 135 из 136 (99,3%) исходных культур и 160 из 163 (98,2%) свежих изолятов. В работе было проведено сравнение тест-систем Crystal™, API® 20Е и VITEK® – сообщается, что из 381 протестированных Enterobacteriaceae, автоматический анализатор правильно идентифицировал 96,1%.
Оценка СЖ-карты относительно Enterobacteriaceae проводилась в 12 лабораториях. Было установлено, что автоматический анализатор позволяет правильно идентифицироватьь 96,7% штаммов Salmonella, 97,0% – Е. coli и 93,0% штаммов других родов бактерий. Междураодная аасоция химиков-аналитиков одобрила применение этого AMS и GWZ-карты в качестве скрининг-метода для предположительной идентификации Salmonella, Е. coli и Enterobacteriaceae, выделенных из пищевых продуктов.
Система VITEK может довольно точно идентифицировать многие микроорганизмы – в нее заложена большая база данных, в любое время можно проанализировать большое количество образцов, а себестоимость одного анализа невелика; вместе с тем стоимость собственно тест-системы довольно высока.
VITEK® 2
При использовании тест-системы VITEK® 2, как и системы VITEK", после первичного выделения культуры микробную суспензию приготавливают в пробиркес солевым раствором и доводят до требуемой густоты с помощью денситометра DensiChek. Затем пробирку с посевным материалом помещают в штатив (кассету). Идентификационный номер образца вводят в смарт-карту с помощью штрих-кода или с клавиатуры, и он автоматически линкуется со штрих-кодом всей тест-карты. Вся поступающая информация переносится в анализатор на микросхему памяти данной кассеты. Это обеспечивает систему отслежиания от ввода данных до составления окончательного отчета.
Сравнение эффективности тест-систем VITEK® Legacy, VITEK® 2 Colorimetric и Phoenix™ (фирмы Becton-Dickinson Diagnostics) при идентификации ферментирующих и неферментирующих бактерий проводилось в работе. Оценивалось в общей сложности 417 изолятов грамотрицательных бактериальных культур, после чего был сделан вывод, что система VITEK® 2 дает правильные результаты на уровне видов без дополнительного подтверждения в 91,7% случаев, и что наиболее точными из всех испытанных тест-систем оказались системы VITEK® 2 и Phoenix™. При использовании дополнительных тестов правильность идентификации у всех трех систем оказалась равна 95%. Наименьший процент ошибочных идентификаций дали системы VITEK® Legacy и VITEK® 2.
В работе было показано, что система VITEK® 2 оказалась также удобной для идентификации дрожжей. С помощью карты VITEK® ID-YST система VITEK® 2 может идентифицировать дрожжи и родственные им микроорганизмы за 15 ч благодаря чувствительной технологии на основе флуоресценции. ID-YST-карта учитывает 47 биохимических реакций; кроме того, база данных этой системы является очень обширна и включает 51 различный таксон. В указанной работе исследовалась надежность VITEK® ID-KST-карты при идентификации дрожжей, обычно обнаруживаемых в клинических образцах. Всего исследовалось 214 штаммов 21 вида дрожжей, изолированных из клинических образцов. Тесты выполнялись на культурах (24-55 ч) с использованием агара Сабуро с гентамицином и хлорамфениколом. Каждый штамм для сравнения параллельно тестировали с помощью стрипов ID 32 С и одновременно проводили микроскопическое исследование клеточной морфологии с тестом на агглютинацию для С. krusei. Идентифицированным однозначно оказались 222 штамма (92,1%), включая 11 изолятов (4,6%), идентифицированных с помощью несложных дополнительных тестов. Еще 10 штаммов (4,1%), для которых результаты оказались неопределенными, невозможно было идентифицировать однозначно даже с помощью дополнительных тестов. Кроме того, 4 штамма (1,7%) оказались идентифицированы ошибочно, а 5 штаммов (2,1%) – вообще не идентифицированными. Тем не менее был сделан вывод, что система VITEK® 2 дает быстрый и точный способ идентификации важных с медицинской точки зрения дрожжей и дрожжеподобных организмов.
11.7.2. Газовый анализ свободного пространства в упаковке
Показано, что газовая хроматография (ГХ) является эффективным способом химической характеризации микроорганизмов. Для идентификации бактерии методом ГХ берут пробу газовой среды из свободного пространства над продуктомс активно растущей микробной популяцией и анализируют ее в целях определения летучих соединений, выделяемых по мере утилизации субстрата ростовой среды. Поскольку различные виды микроорганизмов продуцируют разные побочные продукты метаболизма, полученные хроматограммы можно использовать для их дифференциации. После установления летучих соединений для идентификации микроорганизма используется имеющаяся база данных. Этот метод традиционно использовался для ускоренной идентификации патогенных для человека бактерий. Вместе с тем недавно было полказано, что он может применяться и для оценки запаха, продуцируемого при бактериальной порче мяса птицы. Так как данный метод довольно дорог, его можно рекомендовать лишь для узкого применения в специфических случаях контаминации пищевых продуктов.
11.7.3. Анализ жирнокислотного состава клеток
Микроорганизмы можно также идентифицировать путем экстракции жирных кислот из клеток, их анализа методом ГХ и последующего сравнения полученных профилей с имеющейся базой данных. В течение многих лет анализ низкомолекулярных или летучих кислот использовали для идентификации анаэробных бактерий. Для определения родов и видов бактерий (особенно это касалось неферментирующих грамотрицательных бактерий) использовались жирные кислоты с длиной углеродной цепи в 9-20 атомов углерода. После создания капиллярных колонок из плавленного кварца стало возможным использовать для идентификации многих видов бактерий метиловые эфиры клеточных жирных кислот.
В бактериях обнаружено более чем 300 жирных кислот и родственных им соединений. Фактические данные об их присутствии или отсутствии, а также их количественный анализ можно эффективно использовать для дифференциации видов бактерий. Так, газожидкостная хроматография метиловых эфиров жирных кислот (FAME) оказалась эффективным инструментом для идентификации бактерий, важных в клиническом отношении и при анализе производственной среды пищевых предприятий, причем результаты анализов близки к получаемым в исследованиях по гомологии рибосомальной РНК и ДНК.
В фирме MIDI разработана база данных ММЕ-профилей для идентификации аэробных и анаэробных бактерий и дрожжей. Система Sherlock6 Microbial Identification System данной фирмы включает капиллярный газовый хроматограф Hewlett-Packard, автоматический пробоотборник, детектор, компьютер, принтер, программное обеспечение по идентификации микроорганизмов (MIS) и пакет программ Library Generation Software (LGS) с двумя подпакетами программ кластерного анализа, имеющих возможность «отслеживания» результатов. В этих программах (Dendrogram и 2-D Plot) используются данные, полученные при анализе клеточных жирных кислот микроорганизмов, и в результате получаются простые для восприятия диаграммы изолятов, которые позволяють установить степень их родства с другими микроорганизмами.
Дендрограмма (древовидная диаграмма) создается методом кластерного анализа для генерирования парных совпадений, и в основе ее может лежать анализ кле-точных жирных кислот. Таким образом, она отображает степень родства отдельных штаммов. Этот подход был использован в работе для классификации 368 штаммов Campylobacter spp. (рис. 11.2). По различию жирно-кислотного состава Camp, jejuni и Camp. coli оказались более родственными между собой, чем с Camp, laridis (lari) или с Camp, fetus ss. fetus. Полученная дендрограмма также показала, что у Camp, pyloridis (pylori) эвклидово кодовое расстояние > 30 (53,3) и, следовательно, этот микроорганизм был неправомерно отнесен к роду Campylobacter (в настоящее время его относят к роду Helicobacter). Такой тип аналитической системы прекрасно подходит для целей дифференциации бактерий, однако применяемая для этого база данных нуждается в дальнейшем совершенствовании, причем стоимость оборудования относительно высока.
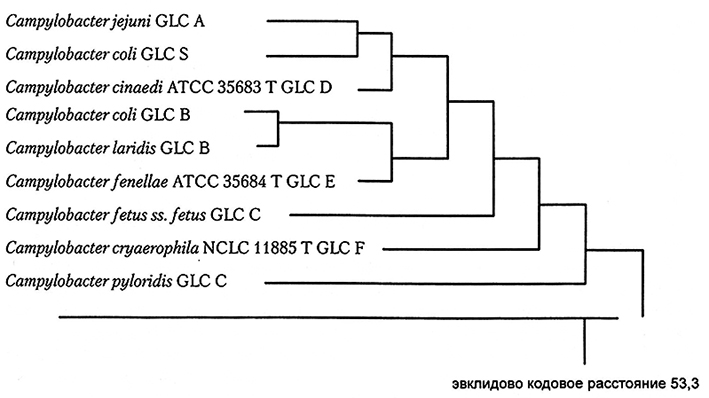
Рис. 11.2. Дендрограмма отдельных штаммов Campylobacter
В работе с помощью системы Sherlock® MIS фирмы MIDI были проанализированы хроматограммы FAМE-изолятов Campylobacter чтобы продемонстрировать присутствие Campylobacter spp. на тушках птицы и в пробах воды из шпарильного чана. На том же самом технологическом оборудовании некоторые штаммы Camp, jejuni вновь появлялись в различные времена года.
11.8. СИСТЕМЫ ТИПИРОВАНИЯ ИЗОЛЯТОВ
ПОСЛЕ ИХ ИДЕНТИФИКАЦИИ
В системах типирования изолятов используется один или несколько методов дифференциации различных штаммов тех или иных бактериальных видов. Применение этих систем необходимо для проведения эпидемиологических исследований и позволяет проследить данный микроорганизм до места его происхождения. Ниже мы рассмотрим некоторые способы типирования, использовавшиеся для разделения видов бактерий на подвиды в прошлом и иногда применяющиеся до сих пор.
11.8.1. Биотипирование
Биотипирование подразумевает дифференциацию подвидов на основе различий в морфологии колоний, метаболической активности и продуцировании токсинов. При использовании этого способа штаммы называют «биотипами». Наиболее широко используемые тесты для диференциации штаммов основаны на способности того или иного микроорганизма сбраживать разные сахара. Как правило, различий по сбраживанию Сахаров бывает недостаточно, и требуются тесты на другие особенности метаболизма. В этих целях разработаны тест-наборы для биотипирования, примером которых могут служит широко доступные API 20Е и Biolog.
11.8.2. Фаготипирование
Бактериофаги представляют собой вирусы, поражающие и разрушающие бактерии. С помощью фаготипирования можно выявить различия между штаммами бактерий путем определения их чувствительности к действию специфических фагов. Штаммовые различия зависят от присутствия или отсутствия определенных рецепторов на поверхности исследуемого бактериального штамма (фаги используют эти рецепторы для прикрепления к клеточной стенке бактерии).
Классическим примером фаготипирования является методика, используемая для Staph, aureus. Для проведения этого теста чистая культура Staph, aureus сеется газоном на поверхность агаровой среды. Затем на поверхность засеянной среды точечно наносят 23 различных фага, о которых известно, что они поражают этот вид стафилококка и по отношению к которым этот микроорганизм демонстрирует штамм-зависимую чувствительность. Затем чашку инкубируют в течение ночи. Стафилококки размножаются до образования видимой биомассы и таким путем образуют бактериальный газон по всей чашке. В зонах, где стафилококки поражаются фагом, клетки лизируются, что приводит к появлению зон просветления в газоне. Профиль чувствительности к фагу служит признаком подвида, так как фаги всегда наносят на чашку в одинаковой последовательности.
11.8.3. Серологическое типирование
Бактериальные антигены находятся на поверхности клетки или жгутика. С помощью специфических антител, присутствующих в специально приготовленной антисыворотке, могут быть выявлены различия между индивидуальными штаммами. Обычно это делается посредством агглютинационных тестов, включая сероагглютинацию впробирке и агглютинацию на предметном стекле. В работе, например, серотипирование выполнялось путем сероагглютинации. Для проведения ориентировочной идентификации использовали поливалентную антисыворотку к Salmonella О (соматические антигены или антигены клеточной стенки) и Я (жгутиковые антигены), а затем с помощью моновалентной антисыворотки была определена дефинитивная антигенная формула (подробно исследование соматических и жгутиковых антигенов описано в работе). Так как антигены О или Н экспрессируются не всегда, в некоторых случаях агглютинации вообще не наблюдается.
11.8.4. Типирование по бактериоцинам
Типирование по бактериоцинам зависит от продуцирования некоторыми штаммами бактерий соединений, которые обычно активны только против других штаммов того же вида. Такие соединения называются «бактериоцины». Тесты проводят в целях определения способности тестового штамма продуцировать один или несколько бактериоцинов со способностью лизировать специфический набор индикаторных штаммов родственных бактерий. Кроме того, можно определить чувствительность тестового штамма к стандартным бактериоцинам. Этот метод используют довольно редко, но прекрасный пример приведен в работе.
11.8.5. Типирование по белкам
Типирование по белкам выполняется для определения белков, продуцируемых различными подвидами бактерий в качестве побочных продуктов метаболизма. Белки экстрагируются из культуры тестового микроорганизма, разделяются электофорезом в полиакриламидном геле (PAGE) и сравниваются с белками, продуцируемыми другими штаммами бактерий. Этот метод относительно недорог, однако по сравнению с другими методами он кажется слишком сложным. В работе сообщается, что при исследовании типирования по белкам относительно Morganella morganii после электрофоретического анализа клеточных лизатов (PAGE-методом) по белкам наружной мембраны молекулярной массой 35-40 кДа штаммы можно было дифференцировать на 21 тип.
11.9. Генетические методы типирования
бактериальных штаммов
Молекулярное субтипирование бактериальных изолятов использовалось в эпидемиологических исследованиях с 1980-х гг. Эти методы включают установление различий между видами или в пределах одного вида путем характеризации клеточных белков или нуклеиновых кислот. В частности, разработаны методы анализа хромосомной ДНК, и молекулярное субтипирование видов в настоящее время считается важным направлением в мониторинге инфекционных заболеваний пищевой природы. Широкое распространение молекулярного типирования привело к созданию большого числа методик и протоколов субтипирования бактерий, с некоторыми из которых мы познакомимся ниже.
11.9.1. Гель-электрофорез в пульсирующем электрическом поле
Применение этой техники применительно к штаммам 5. typhimurium включает разделение ДНК целевой бактерии на сегменты с использованием ферментов рестрикции. Затем молекулы ДНК распределяют в агарозном матриксе, к которому прикладывают электрическое поле. ДНК принимает вытянутую конфигурацию и перемещается по направлению к аноду. Агарозный матрикс содержит крайне нерегулярную сетчатую структуру молекул с порами различных размеров. Так как электрический ток посылается через агарозу импульсами, то в процессе электрофореза миграция ДНК будет зависеть от размера молекул. В традиционном электрофорезе с использоанием постоянного электрического поля молекулы ДНК перемещаются на расстояние, обратно пропорциональное логарифму их длины. Следовательно, у менее крупных молекул относительно небольшое различие в их длине приводит к увеличению различий в их подвижности. Из-за такой логарифмической зависимости чувствительность (разделение молекул в пределах данного матрикса) уменьшается с возрастанием размера ДНК. Кроме того, в случае постоянного электрического заряда крупные молекулы ДНК перемещаются аномально быстро. Для решения этих проблем был разработан метод гель-электрофореза в пульсирующем поле (PFGE), что сделало возможным электрофоретическое разделение более крупных молекул в агарозных гелях. Оригинальный метод подразумевал приложение к гелю пульсирующих переменных ортогональных электрических полей. Таким образом, крупные молекулы ДНК при каждом изменении направления электрического поля останавливались и начинали перемещаться только после переориентации вдоль новой оси поля. Чем больше была молекула ДНК, тем больше времени требовалось для ее переориентации, так что молекулы ДНК с меньшим временем переориентации, чем длительность электрического импульса, можно было разделить на фракции в соответствии с их размерами. Предел разрешения при PFGE-методе зависит от следующих факторов:
- однородность двух электрических полей;
- продолжительность электрических имульсов;
- соотношение продолжительностей импульсов для каждого из переменных электрических полей;
- угол двух электрических полей относительно геля;
- соотношение напряженности двух электрических полей.
В настоящее время используемые для PFGE методы позволяют обеспечить разрешающую способность относительно фрагментов ДНК длиной до 5000 кб*. Получив возможность разделения крупных молекул в агарозе, авторы работы отметили, что метод PFGE расширил диапазон размеров молекул, подлежащих молекулярному анализу и глубоко повлиял на исследование генов и геномов. Для некоторых видов бактерий интактные хромосомы могут быть отделены одна от другой с помощью PFGE, позволяющего использовать Саузерн-гибиридизацию. С помощью PFGE можно обнаруживать полиморфизм длин хромосом, облегчая эволюционные и популяционные исследования бактериальных видов.
________________________
* Килобаза, единица изменения длины молекулы ДНК, равная тысяче пар оснований. - Примеч. перев.
Со всей очевидностью полезность этого метода была продемонстрирована в ходе исследования вспышки пищевых отравлений из-за Е. coli О157 : Н7 в гамбургерах, реализованных через сеть предприятий быстрого питания на западе США в 1993 г. С помощью метода PFGE была проведена характеризация клинических и пищевых изолятов этого микроорганизма и смогли полезность данного метода для отслеживания источников инфекции.
В 1995 г. Центр отслеживания заболеваний (CDС) с помощью Ассоциации медицинских лабораторий (Association of Public Health Laboratories) назначил некоторые государственные лаборатории зональными лабораториями (в сети национального молекулярного субтипирования) по контролю заболеваний, вызываемых бактериями пищевого происхождения. Эта структура позднее стала известна как сеть PulseNet С помощью PFGE эти лаборатории были способны стандартизировать и использовать PFGf-субтипирование и технику анализа профилей, отслеживая и быстро выявляя бактериальное происхождение пищевых заболеваний.
11.9.2. Полимеразная цепная реакция
Полимеразная цепная реакция (ПЦР) представляет собой технику обнаружения присутствия конкретных микроорганизмов в сложной смеси других микроорганизмов на основе амплификации специфических сегментов-мишеней ДНК. В этой реакции в течение нескольких часов образуются миллионы копий специфического сегмента ДНК. Реакционная смесь содержит в 25-100 мкл термостабильную ДНК-полимеразу, свободные нуклеотиды и пару так называемых «праймеров» (коротких ДНК-последовательностей, комплементарных специфической последовательности ДНК-мишени).
Техника ПЦР основана на повторении трех последовательных стадий реакции, каждая из которых имеет место при конкретной температуре. На каждую стадию требуется около 60 с. На первой стадии две цепочки последовательностей ДНК-мишени разделяют путем тепловой денатурации. Как только температура понизится до температуры отжига (45-65 °С), каждый праймер отжигается (гибридизируется) только с одной из разделенных цепочек. Последовательность праймера определяется нуклеотидными последовательностями, фланкирующими последовательность подлежащей амплификации ДНК-мишени. Третьей стадией в этой процедуре является синтез комплементарной цепи на концах каждого праймера. Термостабильная ДНК-полимераза начинает синтезировать новые ДНК-мишени (ПЦР-продукты), присоединяя к праймерам свободные нуклеотиды. Эти вновь синтезированные ПЦР-продукты могут затем служить матрицами в последовательных циклах амплификации. Через 30-40 циклов тепловой денатурации, отжига и увеличения праймеров целевые последовательности ДНК увеличиваются в 109 раз.
Путем такой экспоненциальной амплификации, приводящей к образованию (ПЦР-продукта), где п – число циклов, становится возможным выявить специфические области ДНК путем гель-электрофореза или с помощью компьютеризованных систем обнаружения ДНК. В работе обращено внимание на то, что в некоторых случаях пробы пищевых продуктов, содержащие целые бактерии, можно непосредственно использовать в качестве проб для ПЦР, так как повторяющиеся циклы тепловой денатурации высвобождают бактериальную ДНК. Тем не менее для большинства пищевых образцов при выполнении ПЦР требуется процедура приготовления пробы, так как в них содержатся вещества, способные ингибировать ПЦР, а также вследствие низкой численности патогенных бактерий в продукте.
В работе отмечается, что при обнаружении Chlamydia trachomatis метод ПЦР характеризуется чувствительностью в 99% и специфичностью в 99,7% (чувствительность традиционных методов культивирования составляет только 85,7%, однако их специфичность равна 100%). ПЦР – это крайне чувствительный и специфический метод обнаружения патогенных бактерий в клинических образцах. Что касается образцов пищевых продуктов, применение этой техники все еще затруднено и требует тщательной профессиональной подготовки персонала и обходится недешево, причем этот метод не позволяет различать живые и мертвые бактериальные клетки. Именно поэтому ПЦР не годится для анализа пищевых продуктов после тепловой обработки, исходно содержавших живые патогенные бактерии.
11.9.3. Гибридизация ДНК/РНК
Компания Gene-Trak® Systems, входящая в корпорацию Neogen (Neogen Corporation), разработала аналитические системы на основе ДНК-гибридизации, которые можно использовать для скрининга проб на присутствие Salmonellaf Campylobacter; Listeria, Е. coli, Staph, aureus и Yersinia enterocolitica. Несмотря на то что каждая индивидуальная процедура имеет небольшие различия, ниже мы приведем общее описание этого метода.
Для выполнения анализа на основе ДНК-гибридизации образец сначала необходимо обогатить в неселективной среде, увеличив численность присутствующих целевых микроорганизмов до уровня, при котором они могут быть обнаружены. Для проведения анализа на Salmonella, например, все бактерии в анализируемом образце лизируют с помощью лизирующего раствора, и цепочки целевой рРНК высвобождаются в этот раствор. Затем в раствор добавляют два ДНК-зонда, каждый из которых имеет различные функции. Оба зонда гомологичны к уникальным последовательностям рРНК сальмонелл и гибридизируются к расположенным рядом участкам на одной и той же целевой молекуле рРНК. Иммобилизованный зонд содержит полиадениновый (dA) хвост, который позволяет гибридным молекулам быть захваченными твердым носителем, который связывается с этим хвостом. С помощью флуоресценции детекторный зонд метится с обоих концов. Этот зонд связывается с гибридной молекулой, не связанной с твердым носителем. Таким образом, финальная гибридная молекула содержит цепь рРНК целевой бактерии Salmonella, которая имеет два прикрепленных зонда. Реакция гибридизации выполняется притемпературе 65 °С. Следующим этапом анализа является захват гибрида твердым носителем. Используемый носитель представляет собой пластиковый «дипстик», покрытый поли-dT. Поли-dA-хвост на гибридной молекуле прикрепляется к поли- dT дипстика, и молекула иммобилизуется. Заключительная стадия анализа состоит в обнаружении цепи рРНК Salmonella, иммобилизованной на твердом носителе. Детекторный зонд на другом конце иммобилизованного гибрида перед этим обрабатывают поликлональным антифлуоресцеинантителом (анти-Fl), конъюгированным с пероксидазой хрена (HRP). Этот конъюгат затем связывается с молекулами флуоресцеина на детекторном зонде. Полученный комплекс способен реагировать в присутствии хромогена с субстратом HRP (перекисью водорода). Синий цвет развивается пропорционально количеству конъюгата фермента, связанного с комплексом, и, тем самым, пропорционально количеству иммобилизованой рРНК Salmonella. Реакция останавливается серной кислотой, которая меняет получившуюся окраску с синей на желтую. Интенсивность цвета измеряется путем определения оптической плотности при длине волны 450 нм с помощью фотометра. Оптическая плотность выше предварительно заданных пороговых значений свидетельствует о положительном результате относительно присутствия целевого микроорганизма.
В работе использование ДНК-гибридизации для обнаружения Salmonella в мясе и продуктах из мяса птицы сравнивалось с традиционными методами обнаружения. Сообщается, что процедура ДНК-гибридизации более чувствительна, чем культуральные методы. В результате применения колориметрической ДНК-гибридизации не было получено ни одного ложноположительного или ложноотрицаельного результата. Иная аналитическая система гибридизации, используемая для обнаружения Е. coli, оценивалась в исследовании. С помощью этой аналитической системы были выявлены 233 штамма исследуемой Е. coli, а из 207 штаммов других исследуемых видов лишь ложноположительные результаты дали лишь Е. fergusonii и Shigella. Суммарные ложно-отрицательные результаты дали 1,2% (по сравнению с 23,4% при традиционном культуральном методе обнаружения). В итоге был сделан вывод, что метод гибридизации ДНК значительно более точен, чем традиционные средства обнаружения Е. coli в пищевых продуктах.
11.9.4. Секвенирование последовательности 16S PHK
После открытия ПЦР и секвенирования ДНК сравнение последовательностей генов различных видов бактерий показало, что у каждого вида и среди видов одного и того же рода последовательность 16S рРНК является высококонсервативной и, следовательно, может быть использована как новый стандарт для отнесения микроорганизма к тому или иному виду. На основе этого подхода можно создавать филогенетические деревья, причем бактерии активно переклассифицируют с отнесением их к новым родам. Сообщается об использовании метода секвенирования для идентификации бактериальных штаммов с неопределенными биохимическими профилями, видов бактерий, редко встречающихся в клинических условиях, а также бактерий, не пригодных для культивирования.
11.9.5. Мультилокусный электрофорез ферментов
Для типирования или характеризации бактериальных штаммов в целях систематики и эпидемиологии применяют различные методы – серотипирование, типирование с использованием моноклональных антител, биотипирование, бактериофаговое типирование, типирование на базе фимбрий, типирование по устойчивости к антибиотикам, клеточный электрофорез, электрофорез экстракта всех белков, электрофорез белка наружной мембраны и различные типы углеводоного, липидного и другого химического профилирования и метод снятия «отпечатков пальцев» (фингерпринтинга). Их используют для определения фенотипических вариаций бактериальных видов, однако они дают информацию о частоте аллелей и мультилокусных генотипах, необходимую при анализе генетической структуры популяций бактерий. В крупномасштабных исследованиях по определению генетического разнообразия бактериальных популяций использовался мультилокусный электрофорез ферментов.
Этот модифицированный метод основан на другом, описанном ранее методе изучения млекопитающих. С его помощью изоляты характеризуют по относительной электрофоретической подвижности многочисленных водорастворимых клеточных ферментов. На практике скорость миграции того или иного белка при электрофорезе зависит от суммарного электростатического заряда, а также от аминокислотной последовательности данного белка. В результате можно выделить разновидности ферментов с одинаковой подвижностью, называемые электроморфами или аллоферментами, и сопоставить их с аллелями в соответствующих локусах гена. Сообщается, что профили электроморфов или аллоферментов различных локусов могут быть сопоставлены с мультилокусными генотипами, тогда как число повторностей электроморфов может быть приравнено к повторностям аллелей. Этот метод применяется для различения видов бактерий.
11.9.6. Рибопринтинг
Новая, полностью автоматизированная система характеризации микроорганизмов, называемая RiboPrinter® Microbial Characterization System (фирмы DuPont Qualicon), разработана для описания бактериальных изолятов на основе «отпечатков пальцев» их ДНК. Образец генетического отпечатка пальцев (RiboPrinf) формируется из оперонов рРНК и других близлежащих областей бактериального генома. У бактерий гены рРНК являются высококонсервативными и совершенно уникальными, так что RiboPrint-образцы могут использоваться для различения бактериальных изолятов. Для анализа пробы с помощью этой системы из бактериальных клеток экстрагируют ДНК и фрагментируют ее с помощью фермента рестрикции. Методом электрофореза фрагменты ДНК разделяют по их молекулярной массе и переносят на мембрану, после чего гибридизируют с ДНК-зондом и вводят хемилюминесцентную метку. Гибридизированные таким образом фрагменты флуоресцируют, и изображение получившегося образца фиксируется фотокамерой. Компьютер анализирует изображение образца RiboPrint8 и идентифицирует данную бактерию путем сравнения с имеющейся базой данных.
Риботипирование представляет собой мощный инструмент обнаружения источника инфекции. Например, можно риботипировать бактерию, изолированную из кала инфицированного человека. Риботип затем сравнивают с риботипами тех же видов микроорганизмов, выделенных из различных пищевых продуктов, которые предположительно этот человек мог употреблять в пищу. Подобным образом можно изучать и изоляты от животных или растений, использовавшихся для производства пищевых продуктов, или образцов окружающей среды. Тем самым можно эффективно вычислить источник бактерии, вызывающей инфекцию. Аналогичный подход можно применять и в различных ситуациях производства и кулинарной обработки пищевых продуктов. Очевидно, что преимуществом аналитической системы подобного типа является то, что бактериальные изоляты можно описать глубже видового уровня – только для L monocytogenes получено более 75 различных RiboPrint®-о6разцов. Помимо полезности для обнаружения источников инфицирования человека, эта система может также использоваться для защиты честного имени фирм или предприятий, необоснованно обвиненных в том, что они стали причиной вспышки пищевых отравлений.
Методы риботипирования и PFGE можно совместно использовать для экспресс-идентификации микробных изолятов. В работе автоматическое риботипирование и система PFGE использовались для идентификации 116 штаммов 5. enterica серотипа Newport, в том числе 64 изолятов с устойчивостью ко многим лекарственным препаратам (MDR, multidrug resistant). В результате было успешно проведено различение этих двух групп штаммов с чувствительностью соответственно 100 и 98%, а специфичностью – 76 и 89%. Кластеризация РКг£-фореграмм (но не образцов риботипирования) позволила установить связь между человеческими и коровьими штаммами. Автоматическое риботипирование позволило быстро идентифицировать МDR-штаммы, а гель-электрофорез в пульсирующем поле – обнаружить связи, оказавшиеся полезными в эпидемиологических исследованиях.
Система определения микробиологических характеристик RiboPrint® полностью автоматизирована, но ее стоимость (как и себестоимость одного анализа) очень велика.

