Полимеразная цепная реакция
Методы, основанные на полимеразной цепной реакции (ПЦР) в настоящее время являются одними из наиболее часто использующихся ДНК-технологий анализа генома, в том числе и генома растительного. Метод ПЦР или PCR (от англ. Polymerase Chain Reaction) был разработан К. Маллис с сотрудниками в 1985 г. Суть метода состоит в возможности увеличения количества копий (амплификации) фрагмента ДНК. В основе ПЦР лежат механизмы репликации ДНК и используются аналогичные ферменты репликации. При помощи этого метода можно в миллионы раз увеличить копийность фрагмента ДНК. При этом метод ПЦР основан на использовании специфической ДНК-полимеразы гейзерной термофильной бактерии Thermus aquaticus – Taq-полимеразы. Основное отличие Taq-полимеразы от других ДНК-полимераз состоит в том, что она неинактивируется при нагревании до 95 °С. Температурный оптимум Taq-полимеразы, при котором она синтезирует комплиментарную цепь на ДНК-матрице, находится в районе 68–72 °С. Так же как и другие ДНК-полимеразы, синтетическая активность Taq-полимеразы зависит от наличия одноцепочечной ДНК-матрицы и праймера – небольшой (15–25 н. п.) одноцепочечной олигонуклеотидной последовательности, комплементарной одному из концов ДНК-фрагмента, который необходимо наработать. Поэтому для того, чтобы провести ПЦР реакцию необходимо обязательно знать нуклеотидную последовательность концов амплифицируемого фрагмента. Праймеры могут находить гомологичные области на одноцепочечной матричной ДНК и при определенной температуре реакционной смеси, которая рассчитывается для каждой праймерной пары, гибридизоваться с ними, образовывая двуцепочечные участки. Ферментативная активность Taq-полимеразы, как и у других ДНК-полимераз, активизируется наличием двуцепочечного участка на одноцепочечной ДНК и начинается синтез комплиментарной цепи. Одним из свойств используемой Taq-полимеразы является то, что при ее помощи можно амплифицировать небольшие фрагменты, обычно не превышающие 3000 н. п.
ПЦР проводится следующим образом: в пробирку с реакционной смесью, состоящей из буферного раствора с определенным составом солей, добавляют ДНК-матрицу, дезоксинуклеозидтрифосфаты, два праймера, комплементарных концевым последовательностям фрагмента, который необходимо наработать. ПЦР-реакцию проводят в специальных приборах-термоциклерах. Термоциклеры по сути представляют собой своеобразные нагревательные установки, которые могут быстро (со скоростью более 1 °С/мин) автоматически изменять температуру: нагревать или охлаждать пробирки с реакционной смесью или инкубировать их в течение заданного времени при определенной температуре. При этом термоциклер можно запрограммировать на определенные изменения температурных условий и времени инкубации, а также цикличность повторений этого режима.
Таким образом, каждый цикл состоит из трех основных температурных этапов:
- денатурация ДНК матрицы при 92–96 °С; двуцепочечная ДНК-матрица денатурирует на две одноцепочечные нити. Время этапа денатурации обычно составляет 0,5–1 мин во всех циклах кроме первого. В первом цикле для того, чтобы гарантированно прошла денатурация ДНК-матрицы (обычно геномной ДНК), время этапа денатурации увеличивают до 3–4 мин;
- отжиг праймеров; при так называемой температуре отжига праймера (tотж) праймерные последовательности находят область гомологи й на одноцепочечной матрице ДНК и гибридизуются (отжигаются) с ней, образуя двуцепочечный фрагмент, и являются затравкой для Taq-полимеразы при синтезе комплементарной цепи. Температура отжига индивидуально рассчитывается для каждого праймера в зависимости от нуклеотидного состава и длины. Так как праймеры для конкретного эксперимента определяются непосредственно исследователем, то они обычно подбираются так, чтобы tотж праймера была в интервале температур 30–60 °С. Время этого этапа составляет обычно 15–60 с;
- синтез (наработка фрагмента); при 70–72 °С Taq-полимераза, инициируясь на праймерах, начинает репликацию фрагмента. Эта температура является оптимальной для синтетазной активности Taq-полимеразы. Время этого этапа обычно соотносится с длиной фрагмента, который необходимо наработать и составляет от 0,5 до 2 мин.
При проведении ПЦР для наработки большого количества ДНК фрагмента достаточно 25–30 циклов.
На рис. 2.7 схематично представлены ПЦР-циклы. Разберем гипотетическую задачу: растения табака были трансформированы конструкцией, содержащей маркерный ген nptll, и росли на селективной среде с канамицином. Однако для доказательства трансгенной природы этих растений необходимо показать присутствие векторной конструкции в геноме растений табака. Эта задача решаема с помощью метода полимеразной цепной реакции. Рассмотрим, как будет проходить ПЦР, на примере наработки фрагмента маркерного гена nptll. Для проведения ПЦР необходимо разработать праймеры для проведения конкретной ПЦР и выделить геномную ДНК из анализируемых растений табака. Нуклеотидная последовательность гена nptll известна. Основываясь на этом, были разработаны и искусственно синтезированы праймеры А и В длиной 21 н. п. и 23 н. п. соответственно, комплементарные концам фрагмента, который необходимо наработать. Последовательности праймеров были выбраны таким образом, что tотж их была одинакова и составляла 54 °С.
В пробирке собирают все компоненты для проведения ПЦР, включающую буферную смесь, дезоксирибонуклеотиды, Taq-полимеразу, праймеры А и В и геномную ДНК табака, которая и будет матрицей для ПЦР. Пробирку помещают в запрограммированный термоциклер.
ПЕРВЫЙ ЦИКЛ.
Этап денатурации. При 92–96 °С двуцепочечная ДНК-матрица денатурирует на две одноцепочечные нити (см. рис. 2.7). Как уже было упомянуто, ДНК-мартицей обычно является геномная ДНК и в нашем случае это геномная ДНК табака. Для того чтобы полностью денатурировать геномную ДНК, время денатурации на первом цикле увеличивают до 3–4 мин.
Этап отжига. Температура в термоциклере изменилась и стала 54 °С (до tотж , рассчитанного для праймерной пары А и В). На этом этапе одноцепочечные праймеры А и В находят область гомологий на одноцепочечной матрице ДНК и гибридизуются (отжигаются) с ней, образуя двуцепочечные фрагменты.
Этап синтеза. При температуре в термоциклере 72 °С Taq-полимераза, инициируясь на праймерах, начинает репликацию. В результате на каждой из исходных цепей с каждого праймера синтезируется фрагмент ДНК. При этом синтезированный фрагмент получается значительно длиннее, чем необходимо.
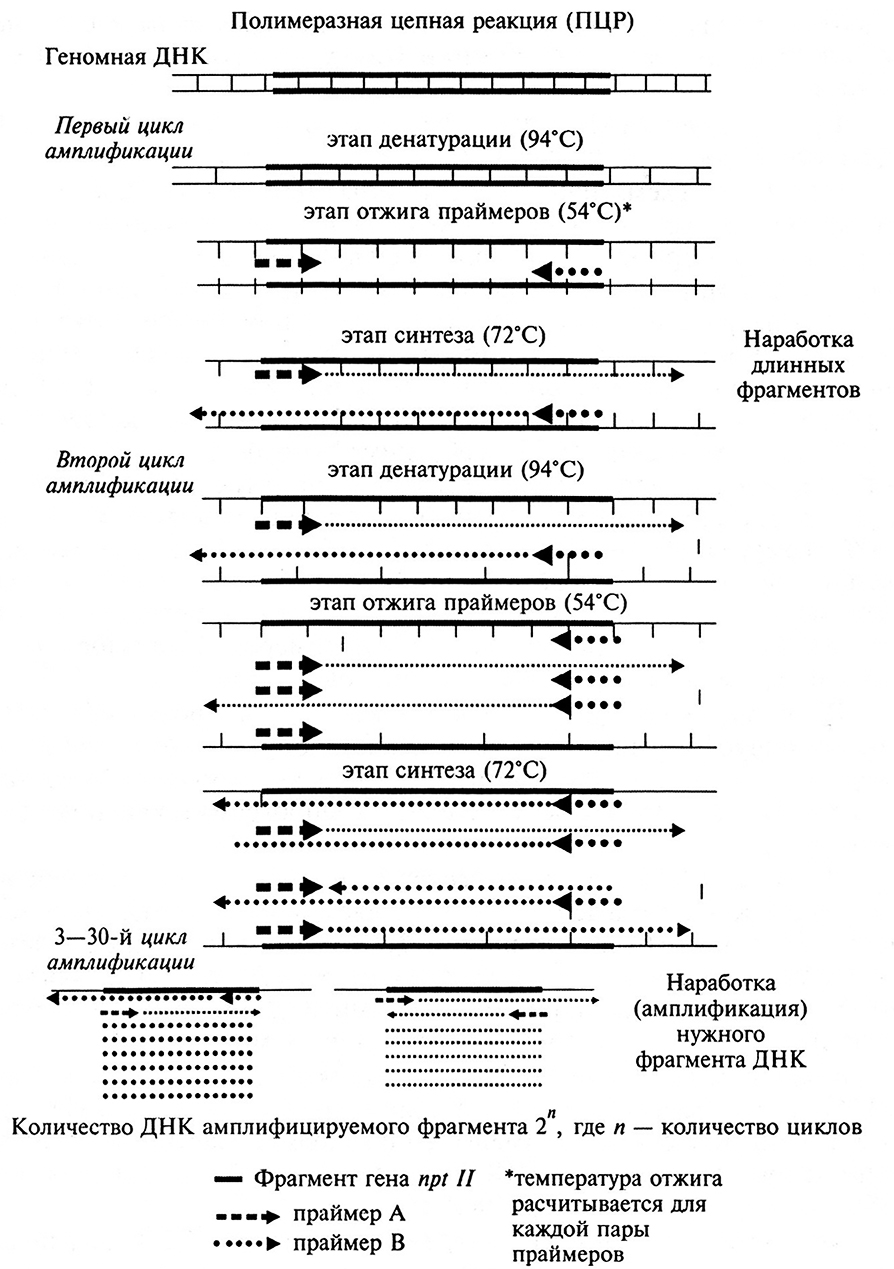
Рис. 2.7. Схема полимеразной цепной реакции
ВТОРОЙ ЦИКЛ.
Изменение температуры до 92–96 °С снова приводит к денатурации нагреванием как исходных, так и вновь синтезированных цепей ДНК. Затем при температуре отжига 54 °С праймеры А и В отжигаются также на всех цепях. При 72 °С Taq-полимераза вновь, используя дезоксинуклеозидтрифосфаты, реплицирует фрагменты ДНК (см. рис. 2.7). При этом на исходных геномных матричных цепях снова образуются длинные фрагменты. Однако когда в качестве матрицы используются длинные фрагменты, синтезированные в предыдущем цикле, то нарабатывается фрагмент необходимой длины, ограниченный по концам праймерами А и В (см. рис. 2.7).
ТРЕТИЙ – 30-й ЦИКЛ.
При 92–96 °С денатурируют все матрицы. При температуре отжига праймеров праймеры А и В, отжигаются на всех матрицах. При температуре синтеза Taq-полимераза реплицирует фрагмент. При этом с каждым циклом число фрагментов nptll гена, ограниченных с двух сторон праймерами А и В, увеличивается в зависимости 2n, где n – число циклов ПЦР. В то время как образование длинных фрагментов будет увеличиваться по линейной зависимости. Таким образом, в ПЦР будет избирательно нарабатываться (амплифицироваться) только необходимый фрагмент ДНК, ограниченный выбранными праймерами. В результате количество полученного фрагмента ДНК в 106 раз превышает исходное количество молекул ДНК-матрицы! После проведения ПЦР полученный фрагмент ДНК детектируется напрямую методом электрофореза в агарозном геле и дальнейшей окраской геля бромистым этидием.
ПЦР – это крайне чувствительный метод анализа. Этим методом можно амплифицировать фрагмент даже в присутствии нескольких пикограмм (10-12 г) ДНК и анализировать ДНК одной клетки. С его помощью можно анализировать фрагменты ДНК, амплифицированные из мумифицированных образцов тканей возрастом более пяти тысяч лет, гербарных образцов, найденных в древних пирамидах, и организмов, замороженных во время Ледникового периода.
В настоящее время полимеразная цепная реакция широко используется в молекулярной биологии и генной инженерии прежде всего для наработки фрагментов ДНК, в большинстве случаев заменяя клонирование в плазмидных векторах. Также ПЦР используют для внесения специфических точковых мутаций in vitro, детекции делеций и инсерций, а также для идентификации специфических или мутантных аллельных вариантов генома, что широко используется для ПЦР-диагностики болезней. ПЦР может быть использована для выделения генов или фрагментов генов из геномных библиотек, для сборки небольших генов из искусственно синтезированных фрагментов. Кроме того, ПЦР используется для секвенирования.
Полемеразная цепная реакция позволяет амплифицировать не только молекулы ДНК, но и РНК. Эта модификация ПЦР-метода называется ОТ-ПЦР (обратно-транскриптазная ПЦР). ОТ-ПЦРсостоит из двух стадий. Первая – получение кДНК и вторая – собственно ПЦР. В результате использование данного метода позволяет амплифицировать ДНК-копии мРНК. Этот метод может быть использован для получения высоко представительных кДНК библиотек из минимального количества исходной мРНК, например кДНК библиотек из клеток трихом или клеток каломеллы. Кроме того, ОТ-ПЦР широко применяют для идентификации РНКовых вирусов или для детекции зараженности организма такими вирусами.
В настоящее время на основе ПЦР разработаны тест-системы для быстрой идентификации огромного числа фитопатогенов растений. В селекции метод ПЦР применяется для анализа гибридного материала, для детекции у потомства определенных генов или определенных фрагментов ДНК, сцепленных с нужным признаком.
Микрочипы. ПЦР-технологии лежит в основе продукции микрочипов (микроматриц), в том числе и экспрессионных микрочипов, позволяющих одновременно изучать работу огромного количества генов. Микрочип представляет собой твердый носитель, на котором в строго определенном порядке иммобилизованы микроколичества известных последовательностей ДНК. В качестве носителя для фиксирования молекул ДНК может быть использован силикон, нейлоновые мембраны или даже специально обработанное стекло. Размеры микрочипов – несколько квадратных сантиметров, на которых нанесены до нескольких тысяч индивидуальных проб ДНК. ДНК для таких проб нарабатывается с помощью ПЦР. Для изготовления микрочипов используют специальные роботы-чипаторы. Образцы ДНК, которые хотят проанализировать при помощи микрочипа, метят ПЦР-методом флуоресцентным красителем и гибридизуют на микрочипе. После отмывки микрочип сканируют при помощи специального лазерного сканера, и данные о флуоресценции каждой точки на микрочипе передаются на компьютер и обрабатываются, используя специальное программное обеспечение. Первый экспрессионный растительный микрочип был получен в 1995 г., когда при помощи ПЦР были получены к ДНК фрагменты 48 генов арабидопсиса и в известном порядке нанесены на микроматрицу. Затем, также при помощи ПЦР были получены два образца: кДНК копии мРНК из корней и из листьев. Эти образцы метили различными флуорометками и гибридизовали с микрочипом. Результаты анализа показали значительные различия в экспрессии анализируемых 48 генов в корнях и листьях. Так были выявлены гены, которые экспрессировались только в корнях или только в листьях, кроме того, для части генов была показана их экспрессия и в клетках корней, и в клетках листьев. Кроме того, были созданы микрочипы, которые можно использовать для сортовой идентификации и паспортизации. Такие микрочипы были созданы на основе SNP-маркеров (маркеров точкового нуклеотидного полиморфизма) для анализа и идентификации сортов картофеля и томатов.
На сегодняшний день создано большое количество всевозможных микрочипов для растений. Так, получены микрочипы для различных тканей растений, определенных семейств генов или генов, вовлеченных в определенные метаболистические процессы. Использование микрочипов является мощнейшим инструментом для анализа механизмов функционирования генома и является одним из перспективных направлений развития нанотехнологий в биологии.
Применение ДНК маркеров в генетике и селекции
Исторически закономерности изменчивости и наследственности изучались при помощи анализа морфологических характеристик. Полиморфизм морфологических признаков и до настоящего времени остается главным критерием в систематике, при изучении мутационного процесса, в селекционных исследованиях филогенетических связей. Основные достижения в селекции до недавнего времени были обусловлены использованием морфологических признаков, на основании которых проводился биометрический анализ. Однако эти признаки имеют ограниченную информативность, далеко не всегда изменяются дискретно, и, как правило, на их проявление существенное влияние оказывают условия возделывания сельскохозяйственных культур. Помимо этого проявляются морфологические признаки чаще всего на поздних стадиях онтогенеза. Кроме того, наблюдается недостаточность морфологических данных при селекции на количественные признаки, такие как продуктивность, качество урожая, устойчивость к биотическим и абиотическим стрессам и др.
Наряду с морфологическими признаками на протяжении многих десятилетий в генетике и селекции широко и успешно используется целый ряд биохимических показателей. Перспективы использования биохимических белковых маркеров в селекции значительно расширились с обнаружением полиморфизма изоферментов и запасных белков. Метод белковых маркеров позволяет решать ряд генетических и селекционных проблем таких, как, например, идентификация сортов злаковых растений, маркирование инбредных линий и оценка их на однородность, отбор ценных генотипов по белковому фенотипу, анализ гибридов, получение информации о подлинности и чистоте партии семян и ряд других прикладных задач. Однако большая часть принятых методов ориентирована лишь на зерновые (пшеница, ячмень, овес, кукуруза) и кормовые культуры (бобовые, некоторые кормовые злаки).
Однако методы белкового маркирования растительных генотипов не лишены целого ряда серьезных недостатков. Прежде всего, поскольку изозимы и белки являются генными продуктами, на степень их экспрессии оказывают влияние факторы окружающей среды, тканеспецифичность экспрессии и различные внутренние факторы, к примеру, возраст растения. Как было показано, даже различия в условиях выращивания и хранения, а также зараженность вирусными заболеваниями могут изменить качественно или количественно белковые спектрограммы растений одного и того же генотипа.
Развитие молекулярной биологии привело к появлению нового класса маркеров – молекулярных маркеров, которые по сути являются фрагментами ДНК. В результате точковых мутаций, вставок, делеций или инверсий гомологичные последовательности ДНК у разных индивидов могут различаться. Такие последовательности ДНК называются полиморфными, а само явление вариабельности нуклеотидного состава гомологичных последовательностей – полиморфизмом ДНК.
В настоящее время полиморфные ДНК используются для маркирования генов, отдельных участков хромосом и целых геномов. Маркирование геномов растений позволяет выявлять степень родства между индивидуальными организмами, что особенно важно при решении прикладных задач, например установление генеалогии сорта или линии для прогнозирования гетерозисного эффекта в программах скрещивания. Еще большее значение молекулярное маркирование геномов растительных организмов приобретает в фундаментальных исследованиях по эволюции и таксономии при популяционном анализе, что имеет прямое отношение к проблеме сохранения биологического разнообразия.
Наибольшее распространение ДНК-маркеры получили также в области молекулярно-генетического картирования геномов различных растений, оценки уровня генетического полиморфизма, при генотипировании особей, линий, популяций, видов. Использование ДНК-технологий во многом позволило преодолеть недостатки метода белковых маркеров в решении ряда таксономических вопросов и отдельных задач селекции, в том числе сертификации сортов. Прежде всего, это связано с тем, что ДНК-маркеры не подвержены влиянию окружающей среды и могут быть идентифицированы на любой стадии развития.
Применение молекулярных маркеров значительно расширило возможности оценки генов устойчивости к болезням и вредителям растений. За короткое время при помощи ДНК-маркеров удалось установить природу устойчивости, в частности, различать моногенную и полигенную резистентность, исследовать взаимодействие соответствующих локусов, определять расовую специфичность отдельных генов, оценивать взаимодействие между генами устойчивости к патогенам.
На данный момент все известные молекулярные маркеры можно условно разделить на три основные группы:
- ДНК-маркеры, основанные на гибридизации (маркеры ПДРФ – полиморфизм длин рестрикционных фрагментов);
- ДНК-маркеры, основанные на использовании принципа полимеразной цепной реакции (RAPD – амплификация полиморфной ДНК с использованием случайных праймеров; маркеры AFLP – полиморфизм длин амплифицированных фрагментов; SSR – микросателлитный анализ и др.);
- ДНК-маркеры, основанные на детекции полиморфизма точковых замен в нуклеотидной последовательности (SNP – точковый нуклеотидный полиморфизм).
Метод ПДРФ-анализа генома. Одним из первых методов, позволивших оценить геномное разнообразие на уровне ДНК, стал метод анализа полиморфизма длин рестрикционных фрагментов (ПДРФ). В основе использования в качестве молекулярных маркеров ПДРФ лежит тот факт, что различные аллели одного локуса могут отличаться по нуклеотидной последовательности. Это могут быть нуклеотидные замены, делеции, вставки или другие перестройки, в результате которых может быть изменен сайт узнавания для какой-либо рестрикционной эндонуклеазы или расстояние между этими сайтами. Таким образом, методом ПДРФ маркируется аллель, расположенный в определенном локусе. Соответствующие аллельные варианты могут быть использованы как маркеры отдельных сегментов хромосом и, соответственно, лежащих в этих сегментах тесно сцепленных генов. Суть метода заключается в гибридизации радиоактивно меченого ДНК-зонда с рестрицированной, разделенной в агарозном геле и перенесенной на нитроцеллюлозный или нейлоновый фильтр геномной ДНК. В результате этого можно детектировать полиморфизм исследуемых геномов, обусловленный как распределением сайтов рестрикции, так и присутствием или отсутствием локусов гомологичных последовательностей ДНК-зонда.
Метод считается высоко воспроизводимым, а получаемые ПДРФ-маркеры – кодоминантными. ПДРФ-маркеры успешно используются как при анализе отдельных участков генома, так и для решения прикладных задач, в том числе для картирования генов и насыщения ДНК-маркерами генетических карт культурных растений, исследования внутривидового разнообразия и выявления филогенетических связей различных систематических групп у растений, а также для паспортизации сортов.
На основе данных ПДРФ-анализа были составлены молекулярно-генетические карты для таких растений, как арабидопсис, рис, картофель, томат, кукуруза, гексаплоидная пшеница, ячмень, горох, люцерна, соя, персик и др. Особенно важно наличие таких карт для различных селекционных программ, так как позволяет определить положение маркируемых агрономически важных генов и признаков и значительно ускорить селекционный процесс. Так, например, у гороха выявлены ПДРФ-маркеры для локуса rb, контролирующего количество липидов в зрелых семенах, и для локуса регуляции экспрессии гена халконсинтетазы. С использованием ПДРФ-маркирования рибосомальной ДНК осуществлена генетическая паспортизация сортов бобов.
Благодаря применению большого набора ПДРФ-зондов появилась возможность сравнивать между собой геномы различных растений, например злаков. Так, при помощи ПДРФ-маркеров показана высокая гомология между локусом Hd-бриса и Vm-A1 пшеницы. Используя общие для пшеницы и ячменя маркеры Xpsr921 и Xmwg634, удалось показать, что гены карликовости ячменя Dwf2 (4HS) и пшеницы Rht-Blc и Rht-Dlc (4BS, 4DS) принадлежат к одной ортологической группе. Подобные исследования позволяют проследить эволюцию хромосом и отдельных генов у важнейших сельскохозяйственных культур.
Метод AFLP-анализа генома. Метод детекции полиморфизма длин амплифицированных фрагментов – AFLP-метод (amplified fragment length polymorphism) представляет собой комбинирование ПЦР- и ПДРФ-методов. Несмотря на техническую сложность, этот метод получил широкое распространение, так как позволяет определять генетические изменения, вызванные точечными мутациями в сайтах рестрикции или делециями/инсерциями внутри рестрикцированного фрагмента. В отличие от метода ПДРФ, AFLP-метод отличается высокой разрешающй способностью и позволяет анализировать одновременно большое количество (до 150–300) локусов генома.
В качестве ДНК-матрицы в AFLP используются рестрицированные двумя различными эндонуклеазами и лигированные с соответствующими адаптерами фрагменты геномной ДНК. После чего гидролизованные ДНК-продукты подвергаются двум последовательным раундам ПЦР амплификации с парой праймеров, гомологичных последовательностям адаптеров и сайтам рестрикций. AFLP-система маркирования считается доминантной, хотя некоторые специалисты отстаивают возможность идентификации и гетерозигот. На практике AFLP-метод широко используется для анализа популяционного полиморфизма, филогенетических отношений, идентификации видов, а также маркирования локусов, сцепленных с различными хозяйственно-ценными признаками. Так, например, был картирован ген Bs2, определяющий резистентность к Xanthomonas campestris pv. vesicatoria у перца, или же локус fadl, обеспечивающий синтез олеиновой кислоты в семенах у озимого рапса.
AFLP-метод также использовался для анализа генетического полиморфизма и эволюционных отношений между современными сортами табака (Nicotiana tabacum) и его дикими сородичами. Отобранные AFLP-праймеры применялись для исследования внутриспецифических различий между местными и современными сортами Vicia sativa L. AFLP-маркеры были использованы в анализе меж- и внутриспецифичных различий 22 природных популяций 13 видов барбариса, произрастающих в Патагонии (Аргентина), с возможностью выявления полиплоидных комплексов. Метод AFLP на сегодняшний день является основным молекулярным методом получения насыщенных молекулярно-генетических карт растений. Карты сцепления на основе AFLP-маркеров были разработаны практически для всех ценных агрономических культур, включая рис, пшеницу, кукурузу, картофель, томат, огурцы, рапс, сахарную свеклу, бананы, лен, люцерну и др.
Кроме того, AFLP-метод также широко используется для маркирования локусов, сцепленных с хозяйственно-ценными признаками. Так были получены AFLP-маркеры, сцепленные с восстанавливающим геном Rfo системы ЦМС у рапса, AFLP-маркеры, позволяющие проводить селекцию на устойчивость к BaYMV у ячменя, дискриминировать нормальные и мутантные аллели Wx-D1 локуса у пшеницы. В околоизогенных линиях перца при помощи AFLP-метода проводилось картирование гена Bs2, определяющего резистентность к бактериальному патогену Xanthomonas campestris pv. vesicatoria. Один из AFLP-маркеров А2 оказался коинтегрирован в локусе Bs2, а два других, F1 и ВЗ, граничат с ним в пределах 0,6 сМ. Сцепленные с геном Bs2 AFLP-маркеры позволили также выявить в локусе мутации, индуцированные гамма-излучением. Локус Gpa2, обеспечивающий нематодоустойчивость картофеля, картирован на хромосоме 12 с использованием информации, основанной на позиционном картировании в геноме картофеля 733 известных AFLP-маркеров. В сегрегационном анализе F2 популяции, полученной от скрещивания двух мутантных линий озимого рапса, при помощи трех AFLP-маркеров удалось картировать локус fad2, обеспечивающий синтез олеиновой кислоты в семенах.
При размножении в культуре ткани ореха пекан (Сагуа illinoinensis) AFLP-анализ использовался для оценки генетической изменчивости сомаклонов, а также меж- и внутрилинейных различий соматических эмбрионов.
Метод RAPD-анализа генома. RAPD (random amplified polimorphic DNA) – метод амплификации полиморфной ДНК, с использованием случайных праймеров аналогично AFLP-методу позволяет исследовать, главным образом, селективно-нейтральные уникальные и умеренно повторяющиеся последовательности генома. Суть метода заключается в использовании для амплификации только одного, обычно 10-нуклеотидного праймера случайной природы, не прибегая к необходимости иметь информацию о нуклеотидной последовательности конкретных участков генома, как в случае классической ПЦР. Метод прост в исполнении, хотя и требует строгой стандартизации условий амплификации. Однако амплифицированные фрагменты «анонимны», их трудно связать с конкретными локусами генома. Кроме того, доминантная природа RAPD-маркеров не позволяет выявлять гетерозиготы.
Благодаря простоте исполнения метод получил весьма широкое распространение и используется для анализа генетического разнообразия различных растительных таксонов, в популяционных и эволюционных исследованиях, а также при построении филогений, решения вопросов систематики растений. RAPD-маркеры широко используются для целей генетического картирования и насыщения генетических карт. Так, этот метод использовался для конструирования карт сцепления в роде Fagopyrum. На основе 596 RAPD-праймеров проведено построение генетической карты сцепления в сегрегационной F2 популяции папайи (Caricapapaya L.), в том числе впервые картирован локус, определяющий пол растения, фланкированный RAPD-маркерами.
Успешное использование RAPD-анализа показано для решения целого ряда спорных таксономических вопросов. Этот метод широко применяется для изучения генетической дифференциации внутри семейства Роасеае и Solanaceae, родов Citrus, Allium, Oriza, Beta и ряда других.
Огромное количество RAPD-маркеров было получено для идентификации локусов, связанных с количественными признаками, так называемыми QTL-локусами. Так были получены RAPD-маркеры, сцепленные с локусом, определяющим засухоустойчивость у ячменя и пшеницы. Было выявлено четыре маркера, сцепленных с повышенной урожайностью в условиях температурного стресса у фасоли. Были получены маркеры QTL-устойчивости к засолению у томата и пшеницы. Было получено большое количество RAPD-маркеров, связанных с QTLустойчивостью к различным болезням растений.
Весьма перспективным оказалось использование RAPD для идентификации сомаклональной изменчивости. Высокая разрешающая способность этого метода позволяет обнаруживать изменения в геноме клеток на разных этапах их культивирования, даже если они не проявляются фенотипически.
Метод ISSR-анализа генома. Относительно недавно стал доступен еще один метод, полученный при комбинировании RAPD-технологии и анализа полиморфизма микросателлитной ДНК. Это так называемое ISSR-маркирование (inter simple sequence repeats – анализ межмикросателлитных последовательностей). Как и RAPD, ISSR не требует предварительного клонирования и секвенирования фрагментов для подбора праймеров. Праймеры представляют собой фрагмент микросателлитного повтора, обычно ди- или тринуклеотидного. ISSR-метод использовался для картирования генома и изучения генетических различий сортов ячменя, насыщения генетической карты сцепления Citrusgrandis, составленной ранее с помощью RFLP, RAPD и изозимных маркеров (Sankar, Moore, 2001).
С целью отбора наиболее ценных генотипов томатов (Lycopersicon esculentum) проводилось картирование гена, обусловливающего этот признак. По результатам анализа сегрегирующих популяций растений с помощью двух ISSR-маркеров на хромосоме 4 томата был картирован локус FgrH, кодирующий высокое соотношение фруктозы к глюкозе в плодах томата и ведущий свое происхождение из дикорастущего вида Lycopersicon hirsutum. Также была показана возможность использования трех ISSR-фрагментов для маркирования признака, сцепленных с мелкозерностью семян гексаплоидной пшеницы, а также четырех ISSR-маркеров, выявляющих признак крупносемянности. Также были идентифицированы ISSR-маркеры, сцепленные с признаком сезонного цветения у Fragaria vesca. Применение ISSR-маркеров также расширило возможности идентификации генов устойчивости к болезням. В качестве примера можно привести картирование кластера генов устойчивости к фузариозному вилту у горошка.
Применение ISSR-анализа позволило определить, к какой из двух субпопуляций риса относится генотип культурного риса, а также с высокой точностью провести сортовую идентификацию внутри каждой субпопуляции. При этом, как показал сравнительный анализ, ISSR-метод характеризуется более высоким полиморфизмом по сравнению с RAPD.
В последнее время ISSR–PCR-анализ используется также для генетической характеристики и поддержания мировых коллекций семян, например таких, как сорта люпина, какао и папайи.
Микросателлиты или простые повторы SSR
(От англ. simple sequence repeats) представляет собой последовательности, состоящие из тандемно повторяющихся 1–6 нуклеотидных единиц. Число повторов в микросателлите и, следовательно, длина самих SSR-последовательностей очень сильно варьирует, что и позволяет их использовать в качестве маркеров. Например, в зависимости от видовой принадлежности растений число микросателлитных повторов на геном колеблется от 5 х 103 до 3 х 105. Микросателлиты присутствуют практически во всех геномах как про-, так и эукариот, и могут быть рассеяны как в гетеро-, так и эухроматиновых областях, включая экзоны, интроны и межгенные участки. Микросателлиты также встречаются в геномах митохондрий и хлоропластов. Из-за своей высокой мутабильности микросателлиты играют существенную роль в эволюции геномов и могут влиять на экспрессию генов.
SSR-маркеры получают на основе ПЦР с использованием двух олигонуклеотидных праймеров, комплементарных уникальным последовательностям, расположенным на концах SSR-локуса. В связи с этим для конструирования праймеров к SSR-последовательностям требуется предварительное получение геномных клонотек, гибридизация их с микросателлитным зондом, секвенирование отобранных клонов и отбор полиморфных микросателлитных локусов.
В последнее десятилетие благодаря высокому уровню вариабельности, кодоминантному типу наследования, простоте детекции с помощью ПЦР, а также необходимости небольшого количества ДНК для анализа микросателлиты стали одними из самых популярных маркеров в исследованиях внутривидового генетического разнообразия, а также генотипирования отдельных образцов. Кроме того, так как микросателлитные последовательности достаточно плотно рассеяны по всему геному, они считаются идеальными генетическими маркерами для построения молекулярно-генетических карт и насыщения карт групп сцепления. К примеру, для риса, несущего в своем геноме от 5 до 10 тыс. микросателлитных копий, удалось выявить 120 независимых микросателлитных SSR-маркеров, маркирующих все 12 хромосом его генома. Для мягкой пшеницы было выявлено 230 микросателлитных маркеров, локализованных на А, В и D геномах. Также проведено генетическое картирование 66 новых микросателлитных SSR-локусов твердой пшеницы. На основе SSR-маркеров была построена генетическая карта сцепления плевела (Lolium perenne L.) при помощи 309 специфичных для данного вида SSR-маркеров. При этом большинство картированных SSR-локусов содержали динуклеотидные (СА)n-повторы. Полученные SSR-маркеры покрывали 54 % генетической карты плевела и преимущественно группировались вокруг предполагаемых центромерных регионов. Подробно охарактеризовано 172 SSR-маркера, использованных при построении молекулярно-генетической карты маниоки. В настоящее время на молекулярно-генетической карте кукурузы маркировано более 1000 микросателлитных локусов. Также генетическая карта сои, включающая 689 RFLP, 79 RAPD, 11 AFLP, 10 изозимных и 26 локусов генетических признаков, была дополнена 606 SSR-маркерами.
Одно из главных применений микросателлитных маркеров связано с возможностью их использования для маркирования и паспортизации сортов, а также разработки диагностических маркеров для важных агрономических признаков. Так, одним из примеров SSR, сцепленного с устойчивостью к болезни, является повтор (АТ)15, локализованный внутри гена белка теплового шока у сои, который располагается на расстоянии 0,5 сМ от гена Rcv устойчивости к вирусу мозаики у сои. Благодаря высокому уровню полиморфизма этот вид маркеров используется для идентификации сортов, анализа внутри- и межпопуляционной изменчивости. Например, с помощью SSR-метода анализировалась генетическая изменчивость различных сортов сои, дыни, персика, черешни, яблони и др. Микросателлитныелокусы были использованы для идентификации близкородственных форм сои (Glycine max и G. soja), характеризующихся низким уровнем изменчивости. Также этот метод использовался для установления генетических различий между видами колосняка – Elymus fibrrosus, Е. alaskanus, Е. caninus и Е. mutabilis (Diaz, 1999), девятью видами рода Licopersicon и некоторыми видами рода Brassica.
Кроме того, микросателлиты имеют огромный потенциал для внутри- и межпопуляционного анализа, предоставляя возможность оценки размеров генетического дрейфа и уровня инбридинга в популяциях. При этом, однако, высокая степень вариабельности микросателлитных локусов, позволяющая различить индивидуальные растения, может ограничить их использование для оценки генетического сходства между менее родственными генотипами. В настоящее время разработаны микросателлитные маркеры более чем для сотни растений, включая наиболее важные сельскохозяйственные виды.
Высокая воспроизводимость SSR-маркеров и их большая дискриминационная способность позволяет использовать их для паспортизации сортов и линий сельскохозяйственных культур. В настоящее время микросателлитные маркеры используются для идентификации сортов картофеля, перца, томата, пшеницы, ржи, ячменя, яблок, роз, олив и др.
STS-метод анализа генома. SCAR-маркеры. В последнее время получил развитие STS-метод, основанный на клонировании и секвенировании фрагментов, полученных в результате RAPD-, RFLP- или AFLP-анализа. Эти короткие последовательности, уникальные для данного генома, используются как маркеры для картирования генов и позиционного клонирования. С помощью STS-маркеров был картирован хлоропластный и митохондриальный геном растений кофе. STS-маркеры, преобразованные из RFLP-фрагментов, были использованы для построения генетической карты ячменя и апельсина. В сегрегационной F2 популяции ячменя на основе RFLP-маркеров хромосомы 3 при помощи STS-метода картировали локусы таких важнейших агрономических признаков, как величина и структура урожая. При сравнении молекулярных карт растений, принадлежащих к различным систематическим группам, при помощи STS-маркеров выявлены гомологичные фрагменты у ячменя и плевела.
STS-маркеры, созданные на основе данных о нуклеотидной последовательности полиморфных RAPD-маркеров, получили название SCAR (sequence characterized amplified region – охарактеризованный секвенированием амплифицированный участок). SCAR-маркеры являются кодоминантными. В этом смысле использование кодоминантных SCAR-маркеров является своеобразным дополнением RAPD-метода и по сравнению с последним имеет несколько важных преимуществ: характеризуется большей информативностью, высокой воспроизводимостью результатов и локусспецифичностью. Также они могут служить точками привязки между физическими и генетическими картами и использоваться для скрининга геномных библиотек.
Используя SCAR-маркеры, удалось осуществить идентификацию и картирование генов устойчивости к вирусу мозаики бобов, пыльной головне у пшеницы и мучнистой росе у гороха. SCAR-анализ позволил маркировать в линиях томата ген устойчивости к вирусу мозаики томата, полученный от дикорастущего вида Lycopersicon peruvianum.На основе RAPD-маркеров, сцепленных с полом, у Actinidia chinensis получены аллель-специфичные SCAR-праймеры, выявляющие нуклеотидные различия между растениями мужского и женского пола, что крайне значимо при проведении селекционных работ. В популяции пшеницы при использовании SCAR-маркера картирован ген устойчивости к листовой ржавчине Lr28, который ведет свое происхождение из Aegilops speltoides. SCAR-маркеры используются для быстрой и эффективной идентификации пола в различных сортах спаржи.
Метод SNP-маркирования. Метод SNP (single nucleotide polimorphism – точковый нуклеотидный полиморфизм) – позволяет исследовать такой наиболее общий тип ДНК-полиморфизма уникальных последовательностей геномов, как точковые мутации. К сожалению анализ SNP в частной генетике растений пока еще не получил такого широкого распространения, как при исследованиях генома человека, хотя, по всей видимости, потенциал этого метода для исследований как в области эволюционной и популяционной генетики, так и для прикладных целей селекции огромен. Относительно недавние исследования SNP-полиморфизма у растений были предприняты пока только при генотипировании образцов кукурузы, картофеля, томата, сосны и сои.
В целом, SNP-анализ может быть полезен для исследования многих биологических феноменов, включая оценку частоты рекомбинации, происхождения популяций, генетического дрейфа. Создание высоко насыщенных генетических карт, видовая, сортовая идентифккация, а также характеристика коллекции генбанков представляют, еще одну обширную область применения этого метода.
SNP имеют относительно низкую мутационную скорость по соединению с микросателлитными локусами, и, как правило, биаллельны. Однако комбинированные гаплотипы могут быть идентифищррваны посредством анализа тесно сцепленных SNP-аллелей. В Настоящее время разработано множество методов детекции SNP, включая аллель-специфичный PCR, TaqMan PCR, аллель-специфичную гибридизацию, пиросеквенирование, гибридизацию на микрочипах и другие техники, применение которых определяется целыми и задачами конкретного исследования.
2.16. НЕРЕШЕННЫЕ ПРОБЛЕМЫ ГЕННОЙ ИНЖЕНЕРИИ РАСТЕНИЙ
При значительном прогрессе в генной инженерии растений остается большое количество нерешенных задач. Одна из проблем связана с трудностью одновременного введения в геном растений больших (более 10 т. н. п.) генов или нескольких функциональных генов. Это связано с емкостью векторов для трансформации. Сами гены, особенно эукариотические, которые в последнее время все чаще используются для трансформации растений, значительны по размерам (5–15 т. н. п.). Но кроме выбранного гена векторные конструкции должны иметь в своем составе селективные гены. В некоторых случаях для уменьшения длины используют кДНК-последовательность гена. Однако кДНК-копии не всегда приемлемы из-за специфики сплайсинга in vivo или из-за присутствия, обычно в первом интроне, специфических регуляторных последовательностей. Лимитирующим фактором при использовании для трансформации растений может быть и то, что необходимый признак кодируется несколькими генами и получение трансгенных растений, обладающих таким признаком, в настоящее время технически несколько затруднено.
Отдельно стоит проблема, возникающая при экспрессии чужеродного гена: часто после двух-пяти поколений активно транскрибирующийся трансген перестает экспрессироваться. Сточки зрения логики это понятно: растительная клетка активно (например, из-за сильного конститутивного промотора 35S CaMV) экспрессирует чуждый для ее метаболизма белок, обычно бактериального происхождения. Чаще всего инактивация трансгена происходит из-за метилирования регуляторных последовательностей либо возможна репресссия в результате взаимодействия с промотором каких-то белков. Активизировать такой «выключенный» трансген не представляется возможным. Спрогнозировать, будет ли со временем проходить такое «молчание» трансгена довольно трудно, так как оно зависит от ряда факторов, в том числе, по всей видимости, от последовательности самого белка и конкретного места интеграции его в геном растения. Преодолеть это, вероятно, в какой-то мере, возможно путем получения многократной трансформации и получения различных линий, несущих одинаковую векторную конструкцию с различными местами интеграции в геном растений.
Одной из главных причин, сдерживающих интенсивность и эффективность работ по трансгенозу, остается крайне слабое развитие исследований по идентификации эффективных генов, созданию банков генов и ограниченная научная база генетической инженерии, что связано с крайне слабой финансовой поддержкой в нашей стране биоинженерии как важнейшего приоритета XXI в.

